使用 CRISPR,我们能够识别并快速地改变目标基因、诊断疾病、预测个体的疾病易感性,应用 CRISPR 的农业产物已经走进现实,CRISPR 迈出进入临床医学的第一步已然不远……曾几何时,人类基因组测序需 5 年才能完成;如今,24 小时内获取完整序列已经变得可行。
CRISPR 是一门年轻的技术,它何以具有如此之大的能量?
近期,因开发 CRISPR 基因编辑技术于2020年获得诺贝尔化学奖的 Jennifer Doudna 在《科学》杂志撰文[1],回顾了过去十年中 CRISPR 基因编辑的起源和应用、当前成果、局限性,并讨论了未来的发展方向。就让我们跟随这篇文章一起来看看 CRISPR 的腾飞之旅。

论文题图
改变生物学的技术
1987年,一篇论文首次将 CRISPR 带入人类的视野[2]。CRISPR 是在微生物基因组中发现的一组规律重复的基因序列,它常与编码 CRIPSR 相关蛋白(Cas)的基因一同出现。有趣的是,这些短重复序列竟然与病毒中的部分序列匹配[3]。
原来,CRISPR 系统其实是微生物的一种自我防御系统,通过 CRISPR 转录产物识别、Cas 蛋白切割来破坏病毒的 DNA 或 RNA,以此防御病毒入侵[4]。而这一运作系统的简单和精巧使其具有了成为基因编辑工具的可能。
目前使用最广泛的是被认为是第三代基因编辑技术的 CRISPR-Cas9 系统,由携带的引导 RNA(guide RNA)来识别序列,Cas9 蛋白(一种 RNA 指导的 DNA 内切核酸酶)切割 DNA,因此可以广泛地靶向不同的序列。通过改变 Cas9 蛋白活性位点的氨基酸,也可以仅切断 DNA 单链或者仅结合不切割,由此实现转录抑制、激活、沉默、上调等多种基因调控目的[5]。对 Cas 蛋白的进一步改造,也提供了单碱基编辑、染色质修饰、序列插入、靶向 RNA 等多样的应用方向[6]。
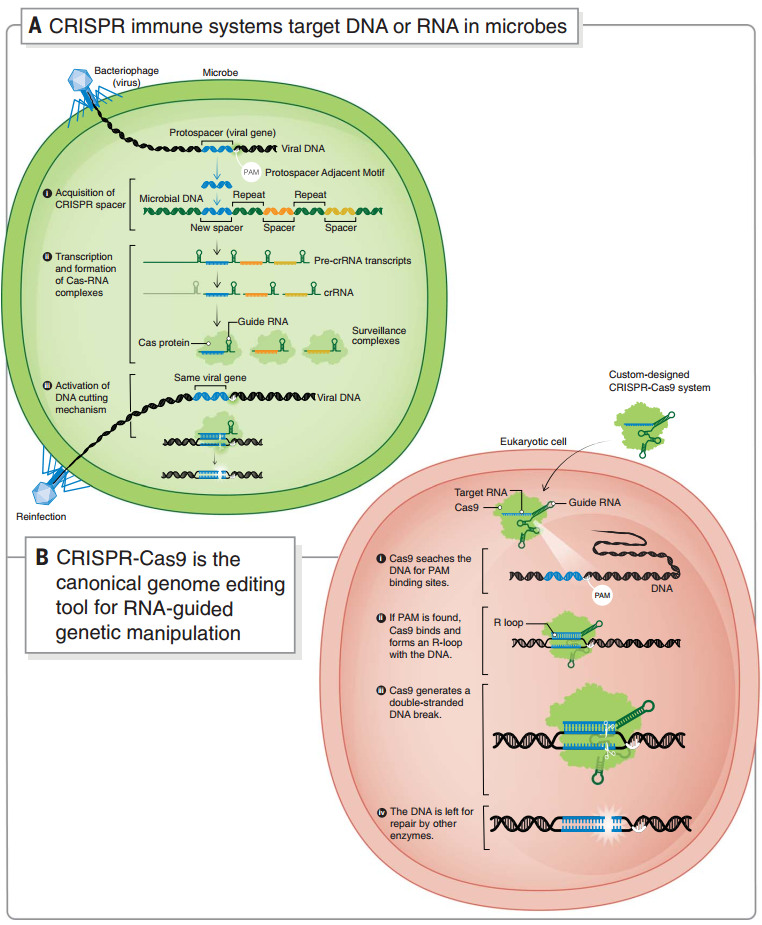
微生物中的 CRISPR 自我防御系统和 CRISPR-Cas9 基因编辑系统
CRISPR 技术的出现为多种疾病治疗提供了基础,包括镰状细胞病(SCD)、乙型地中海贫血、转甲状腺素蛋白(TTR)淀粉样变性、先天性眼病等遗传疾病,乃至癌症、艾滋病毒感染等常见病;CRISPR 技术已经在农作物和家畜育种方面取得成果,比如更耐热应激的奶牛(slick-coat cattle)及更有营养的番茄;而在基础研究上,CRISPR 作为有力的科研工具,加速了分子和细胞生物学领域的研究,在数千篇论文中起到了作用。
接下来,就让我们来具体了解一下 CRISPR 这个优秀的科研工具箱。
“万能”工具箱
CRISPR 结构精巧灵活、可塑性极强,不同 CRISPR 体系具有多种功用,接下来我们选取其中使用较为广泛的三类进行介绍。
①基因敲除
基因敲除是 CRISPR 最主要的应用之一,真核细胞中普遍使用 CRISPR-Cas9 系统,由工程单导 RNA(sgRNA)将 Cas9 核酸酶引导到目标位点,制造 DNA 双链断裂(DSB),由非同源末端连接(NHEJ)和同源定向修复(HDR)等内源性修复途径修复。
这种技术已经广泛应用于基因敲除动物模型的创建,由于绕过了传统手段的复杂胚胎干细胞筛选过程,培育时间大大缩短,培育转基因小鼠的时长从 1 年缩短到了 4 周[7]。同时,大多数哺乳动物并没有成熟的胚胎干细胞系,通过 CRISPR-Cas9 也能够促进新物种的动物模型出现。
在生殖细胞编辑之外,CRISPR-Cas9 也可以用于体细胞编辑,这避免了部分基因全身敲除的致命影响,在特定场景下也更有利于模拟癌症的演变和进展以及确定癌症治疗的新策略[8]。
利用 CRISPR,研究者已经制造了多种疾病的动物模型,包括酪氨酸血症、杜氏肌营养不良、癌症、骨质疏松、亨廷顿舞蹈症、肌萎缩型侧索硬化症、阿尔茨海默病和艾滋病等[9]。
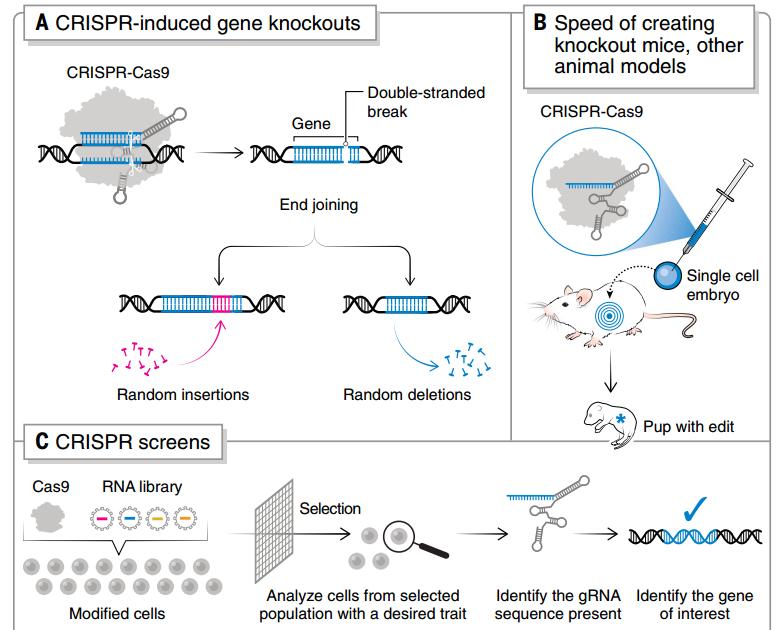
CRISPR 敲除和 CRISPR 筛选的流程
② CRISPR 筛选
当通过 CRISPR 并行执行针对多个基因的遗传改变,就是 CRISPR 筛选。由于灵活高效,CRISPR 可以方便地引入基因扰动,可以深入研究单个基因扰动如何影响目标细胞的功能,又或是在混合筛选中对数千个扰动进行高通量测序[10]。当 gRNA 文库达到基因组规模,研究者可以轻易使用 CRISPR 来扰动人类基因组中的每一个基因[11]。
在 CRISPR 敲除(CRISPR KO)筛选之外,CRISPR 干扰(CRISPRi)筛选和 CRISPR 激活(CRISPRa)筛选也是常用的可逆的控制基因表达的筛选方法[12];饱和基因组编辑(saturation genome editing)能够产生所有可能的单核苷酸多态性(SNPs)用于功能筛选[13];后文会介绍的单碱基编辑技术能够以更高效率引入点突变,或许会成为 CRISPR 筛选的新助力。
CRISPR 筛选已经在类器官、动物、植物等复杂模型中实际应用,它目前是分析癌基因功能的常用方法,还可以用于识别各种癌症驱动因素和调节因素[10]。
结合其他技术,CRISPR 筛选同样可用于细胞表型筛选、肿瘤耐药性筛选等等领域,比如 Procode、Perturb-ATAC、Perturb-seq 和 ECCITE-seq 等多组学筛选技术。
③单碱基编辑技术
单碱基编辑技术提供了一种在不切断 DNA 双链的情况下进行基因编辑的新可能,这一方面可以产生精确的点突变,一方面无需复制模板且限制了多余的副产物。
单碱基编辑系统通常由不具有催化 DSB 活性的 dCas9、dCas12a、dCas13b 等 Cas 蛋白和碱基脱氨酶融合组成,sgRNA 将融合酶引导到目标位点,脱氨酶进行化学修饰,通过细胞修复机制处理碱基不匹配。在过去的几年里,DNA 和 RNA 单碱基编辑已经实现了 C>T、A>G、C>G、A>I和C>U 转换突变。
点突变是人类致病遗传变异的主要类别,单碱基编辑技术为治疗人类遗传疾病铺平了道路。单碱基编辑已经在多种小鼠模型中表现出了纠正功能丧失突变的良好效果,例如体内单碱基编辑纠正小鼠 Hutchinson-Gilford 早衰综合征[14]。使用单碱基编辑技术治疗家族性高胆固醇血症的早期临床试验已经开始,近期将开展的还有针对镰状细胞病的临床研究[15]。
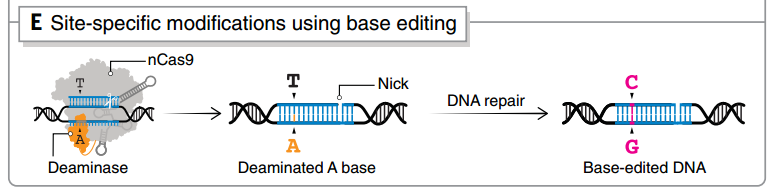
单碱基编辑技术之一base-editing的原理
CRISPR 进化的方向
随着 CRISPR 基因编辑技术进入下一个十年,还有一些关键的问题需要解决,其中一个就是编辑的精度。这既指编辑目标位点的特异性,也指编辑结果的准确性。
为减少意外结合和切割导致的脱靶效应,研究者利用合理的设计开发了高保真Cas变体,例如SpCas9-HF1、evoCas9、HiFiCas9和 Cas9_R63A/Q768A 等变体,亦或是设计了优化的导向方法,例如 E-Crisp、CasOFFinder、sgDesigner。近期,也有研究开发了机器学习工具来预测基因编辑结果,但目前尚未得到体内研究的验证[16]。
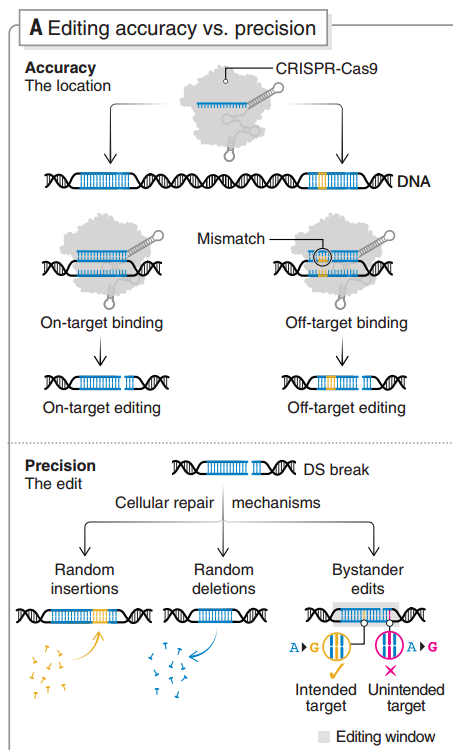
基因编辑特异性和准确性
传统编辑中,DSB 后涉及 NHEJ 或 HDR 通路的修复(前者容易出错,可能导致突变,后者虽更精确,但发生率非常低),这会导致一系列插入缺失突变(indels)。避免这个问题的其中一个思路就是提高 HDR 效率和/或抑制NHEJ,例如使用单链寡脱氧核苷酸模板[17]、通过控制细胞周期阶段来促进 HDR [18]、使用位点特异性 Cas9 寡核苷酸偶联物将 DNA 模板募集到目标位点[19]。但在这些方法之下仍存在 DSB 相关的大量基因位点缺失和染色体重排风险,可能导致基因组不稳定。
避免产生DSB则是另一个思路,碱基编辑(base editing,BE)和引导编辑(prime editing,PE)是目前的主要方法。不过 BE 也存在旁观者编辑的问题,当编辑窗口中存在多个目标碱基时,BE 的编辑精度会大大降低[20]。Cas 蛋白识别特定原间隔序列邻近基序(PAM),受此限制,缩小编辑窗口也存在一定困难,这还需要结合多种辅助策略来改善。PE 同样不涉及 DSB,且能够实现 DNA 序列的插入,但目前 PE 的编辑效率还比较低[21]。
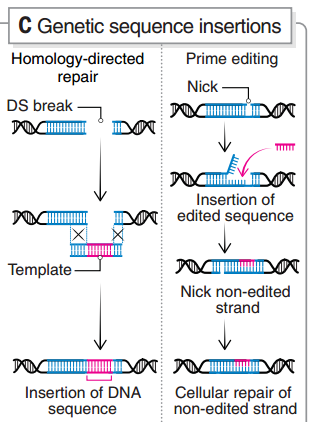
引导编辑的原理
限制 CRIPSR 应用的另一个环节是 CRISPR 的递送。目前在哺乳动物系统中存在多种递送方法,可以大致分为物理递送、病毒递送和基于合成材料的递送。常用的物理方法包括微注射和电穿孔,传递效率高且剂量可控,但这局限于体外递送;病毒递送常用腺相关病毒(AAV)、腺病毒(AdVs)和慢病毒等载体,其中 AAV 在临床研究中应用较多,但病毒载体存在载量、免疫原性、制造成本等多种潜在问题;脂质纳米颗粒(LNPs)、阳离子聚合物、多肽和纳米金(gold nanoparticles,GNPs)等合成材料载体通常较为安全可控,LNPs 是首次成功用于治疗TTR淀粉样变性的递送载体[22]。
体内环境对 CRISPR 递送具有多种阻碍,载体需避免降解、调理素作用、吞噬导致从血管渗出,有效穿过狭窄间隙,并在内吞时释放内含物。CRISPR 治疗人类疾病能够发展到何种程度,很大程度上将取决于当前递送方法的改进、新递送方法的创建和设计更紧凑的 CRISPR 编辑系统。
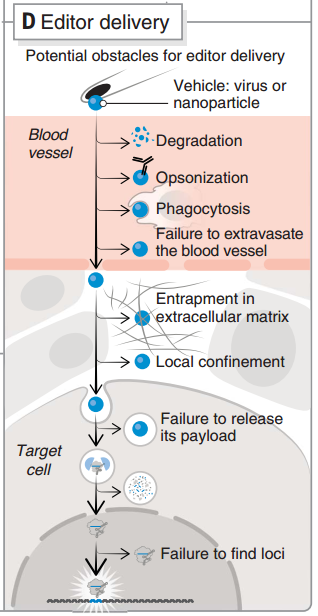
CRISPR 递送过程
结语
可编程基因编辑技术的出现为细胞和基因治疗的应用铺平了道路。已有大量应用 CRISPR 的临床试验正在进行,其中关于 SCD 的至少已有 8 项正在进行或即将开始,预计 2023 年内 FDA 会启动至少一项疗法的批准[23]。随着临床应用的扩大,CRISPR 也有可能成为预防神经退行性疾病和心血管疾病的一种手段[24]。
CIRSPR 也对农业和畜牧业产生了深远的影响,CRISPR 编辑的食品已经进入市场,包括营养价值更高的番茄和两种已获批在日本销售的鱼类。CRISPR 精准编辑的特点非常适合定向育种,可以同时敲除和激活不同的基因,例如给小麦引入抗病性的同时增加小麦的产量[25]。
CRISPR-Cas9 和 CRISPR-Cas12a 是目前应用最广的编辑系统,已经在全世界各地的实验室得到了广泛的应用。这加速了基础研究的进展,也倒推了 CRISPR 工具箱的扩展。结合机器学习、活细胞成像、DNA 测序技术, CRISPR 还能做到更多。
下一个十年,CIRSPR 还将继续改变世界。
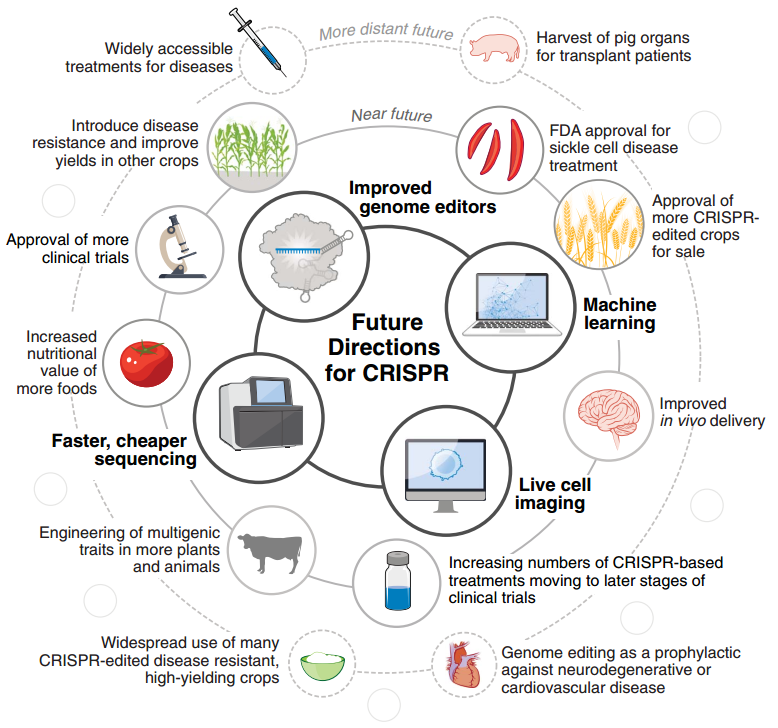
未来的 CRISPR
本文转载自“泰格医药”微信公众号。
参考资料:
[1] Wang J Y, Doudna J A. CRISPR technology: A decade of genome editing is only the beginning[J]. Science, 2023, 379(6629): eadd8643.
[2] Y. Ishino, H. Shinagawa, K. Makino, M. Amemura, A. Nakata,Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. J. Bacteriol. 169,5429–5433 (1987). doi: 10.1128/jb.169.12.5429-5433.1987;pmid: 3316184
[3] F. J. M. Mojica, C. Díez-Villaseñor, J. García-Martínez, E. Soria,Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J. Mol. Evol.60, 174–182 (2005). doi: 10.1007/s00239-004-0046-3;pmid: 15791728
[4] S. J. J. Brouns et al., Small CRISPR RNAs guide antiviral defense in prokaryotes. Science 321, 960–964 (2008).doi: 10.1126/science.1159689; pmid: 18703739
[5] L. S. Qi et al., Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression.Cell 152, 1173–1183 (2013). doi: 10.1016/j.cell.2013.02.022;pmid: 23452860
[6] J. Y. Wang, P. Pausch, J. A. Doudna, Structural biology of CRISPR-Cas immunity and genome editing enzymes.Nat. Rev. Microbiol. 20, 641–656 (2022). doi: 10.1038/s41579-022-00739-4; pmid: 35562427
[7] H. Yang, H. Wang, R. Jaenisch, Generating genetically modified mice using CRISPR/Cas-mediated genome engineering. Nat. Protoc. 9, 1956–1968 (2014). doi: 10.1038/nprot.2014.134; pmid: 25058643
[8] H. Mou, Z. Kennedy, D. G. Anderson, H. Yin, W. Xue, Precision cancer mouse models through genome editing with CRISPRCas9. Genome Med. 7, 53 (2015). doi: 10.1186/s13073-015-0178-7; pmid: 26060510
[9] H. Lee, D. E. Yoon, K. Kim, Genome editing methods in animal models. Anim. Cells Syst. 24, 8–16 (2020). doi: 10.1080/19768354.2020.1726462; pmid: 32158611
[10] A. Katti, B. J. Diaz, C. M. Caragine, N. E. Sanjana, L. E. Dow,CRISPR in cancer biology and therapy. Nat. Rev. Cancer 22,259–279 (2022). doi: 10.1038/s41568-022-00441-w;pmid: 35194172
[11] N. E. Sanjana, O. Shalem, F. Zhang, Improved vectors and genome-wide libraries for CRISPR screening. Nat.Methods 11, 783–784 (2014). doi: 10.1038/nmeth.3047;pmid: 25075903
[12] L. Przybyla, L. A. Gilbert, A new era in functional genomics screens. Nat. Rev. Genet. 23, 89–103 (2022). doi: 10.1038/s41576-021-00409-w; pmid: 34545248
[13] G. M. Findlay et al., Accurate classification of BRCA1 variants with saturation genome editing. Nature 562, 217–222 (2018).doi: 10.1038/s41586-018-0461-z; pmid: 30209399
[14] L. W. Koblan et al., In vivo base editing rescues Hutchinson-Gilford progeria syndrome in mice. Nature 589, 608–614 (2021). doi: 10.1038/s41586-020-03086-7;pmid: 33408413
[15] K. Kingwell, Base editors hit the clinic. Nat. Rev. Drug Discov.21, 545–547 (2022). doi: 10.1038/d41573-022-00124-z;pmid: 35831515
[16] W. Chen et al., Massively parallel profiling and predictive modeling of the outcomes of CRISPR/Cas9-mediated doublestrand break repair. Nucleic Acids Res. 47, 7989–8003(2019). doi: 10.1093/nar/gkz487; pmid: 31165867
[17] C. D. Richardson, G. J. Ray, M. A. DeWitt, G. L. Curie,J. E. Corn, Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nat. Biotechnol. 34, 339–344(2016). doi: 10.1038/nbt.3481; pmid: 26789497
[18] A. Lomova et al., Improving gene editing outcomes in human hematopoietic stem and progenitor cells by temporal control of DNA repair. Stem Cells 37, 284–294 (2019).doi: 10.1002/stem.2935; pmid: 30372555
[19] X. Ling et al., Improving the efficiency of precise genome editing with site-specific Cas9-oligonucleotide conjugates.Sci. Adv. 6, eaaz0051 (2020). doi: 10.1126/sciadv.aaz0051;pmid: 32494588
[20] M. Arbab et al., Determinants of base editing outcomes from target library analysis and machine learning. Cell 182, 463–480.e30 (2020). doi: 10.1016/j.cell.2020.05.037; pmid: 32533916
[21]M. M. Hassan, G. Yuan, J.-G. Chen, G. A. Tuskan, X. Yang,Prime editing technology and its prospects for future applications in plant biology research. BioDesign Research 2020, 1–14 (2020). doi: 10.34133/2020/9350905
[22] J. D. Gillmore et al., CRISPR-Cas9 In vivo gene editing for transthyretin amyloidosis. N. Engl. J. Med. 385, 493–502(2021). doi: 10.1056/NEJMoa2107454; pmid: 34215024
[23] F. D. Urnov, The Cas9 hammer-and sickle: A challenge for genome editors. CRISPR J. 4, 6–13 (2021). doi: 10.1089/crispr.2021.29120.fur; pmid: 33616446
[24] K. Musunuru et al., In vivo CRISPR base editing of PCSK9 durably lowers cholesterol in primates. Nature 593,429–434 (2021). doi: 10.1038/s41586-021-03534-y;pmid: 34012082
[25] S. Li et al., Genome-edited powdery mildew resistance in wheat without growth penalties. Nature 602, 455–460(2022). doi: 10.1038/s41586-022-04395-9;pmid: 35140403







