导读:来自华盛顿大学的研究人员发表文章,报道了第三代DNA测序技术新机制——纳米孔检测核酸碱基数增加至20-30个,这将激化目前纳米孔测序技术市场竞争,相关成果公布在Nature Biotechnology杂志上。
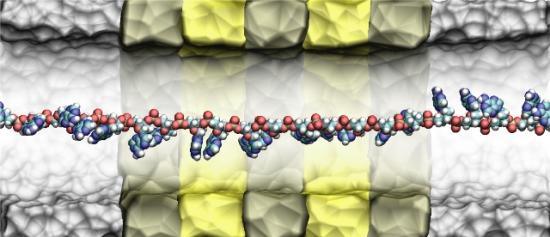
纳米孔检测核酸碱基数增加至20-30个
来自华盛顿大学的研究人员发表了题为“Reading DNA at single-nucleotide resolution with a mutant MspA nanopore and phi29 DNA polymerase”的文章,报道了第三代DNA测序技术新机制——纳米孔检测核酸碱基数增加至20-30个,这将激化目前纳米孔测序技术市场竞争,相关成果公布在Nature Biotechnology杂志上。
文章的通讯作者是华盛顿大学的物理学家Jens H.Gundlach教授,他在纳米孔测序技术方面成就赫然,曾经设计出了一种可以在纳米孔内对DNA进行快速测序的新方法,这种纳米微孔只有1个纳米大小,仅够用来测量一个DNA的单分子链。
对于这一最新成果,他表示,“为了能获得可行的,易于操控的测序平台”,“我们提高了一种蛋白纳米孔的效用,这种纳米孔能与分子马达共同作用,帮助DNA链每次通过一个核苷酸。”
另外一种思考方向
正如前文所叙,纳米孔测序技术存在瓶颈——DNA链通过纳米孔的速度太快,以致来不及对碱基进行检测,因此科学家们想尽办法降低这一速度。Gundlach教授研究组这一成果就是朝着这个方向,但是也有其它的研究组想到了另外的方法。
比如近期来自哥伦比亚大学电气工程教授Kenneth L Shepard就研发出一种比目前市面上仪器精确度更高的分析方法。并且研究人员利用商业化半导体技术,设计了一种定制集成电路,构建新型放大芯片纳米检测设备,可以缩小仪器的体积,用于医学应用。正如研究人员所说的那样,“大部分研究组都尝试减缓DNA的速度,而我们的方法却是构建更快的检测设备”,“我们将最灵敏的电子设备,与最灵敏的固态纳米孔结合起来了。”
目前许多单分子检测都是通过光学技术完成的,这些技术利用荧光分子在特殊波长中发射光子,但是当荧光非常强的时候,就会出现一个主要问题,那就是每个分子通常每秒只会生成几千个光子,这也就是说,无法观测到比毫秒速度更快的事件,因为更快速度成像的图片会太暗了,相反,如果你可以采用电子或者离子检测方法,那么就能在一秒钟内获得上十亿个信号。但电子检测方法的问题就在于没有能与荧光波长过滤器匹配的设备,因此即使信号产生了,也常常被掩盖在背景噪音中。
这一纳米孔检测设备十分简单,而且易于携带,它不需要完整的显微设备,也不需要高功率仪器,唯一需要注意的就是实验细节。不难想象在未来几年里,纳米孔成像技术将对DNA测序,以及其它医学应用产生巨大的影响。
第三代测序市场产品
第三代测序技术则是基于纳米孔的单分子读取技术,这种方法读取数据更快、有望大大降低测序成本,改变个人医疗的前景。这一技术的研发是系统工程,涉及生物、半导体、计算机、化学、光学等多个领域,需要不同学科顶尖力量的合作。目前市场上已经出现了相关产品。
比如近期Oxford Nanopore Technologies(以下简称Oxford)发布了两款DNA测序仪,高通量的GridION单元和U盘大小的便携式MinION单元。
Gordon Sanghera现任Oxford Nanopore的CEO。他表示:“我们设计的MinION和GridION系统具有一系列新特性。纳米孔非常适合简单的的样品制备和新型的信息学流程,系统可设置为运行实验,直到收集了足够数据,才来总结实验。”Oxford Nanopore希望到2013年,整个人类基因组的测序能在15分钟内完成。那将需要20个GridION节点,每个带有8000个纳米孔。
除此之外罗氏也准备开发相关产品,他们去年从亚利桑那州立大学的Biodesign研究所和哥伦比亚大学纳米科学中心获得了一些技术授权,以开发一种全新的DNA测序系统。

 Reading DNA at single-nucleotide resolution with a mutant MspA nanopore and phi29 DNA polymerase
Reading DNA at single-nucleotide resolution with a mutant MspA nanopore and phi29 DNA polymerase
Elizabeth A Manrao,Ian M Derrington,Andrew H Laszlo,Kyle W Langford,Matthew K Hopper, Michael Niederweis& Jens H Gundlach
Nanopore technologies are being developed for fast and direct sequencing of single DNA molecules through detection of ionic current modulations as DNA passes through a pore's constriction1, 2. Here we demonstrate the ability to resolve changes in current that correspond to a known DNA sequence by combining the high sensitivity of a mutated form of the protein pore Mycobacterium smegmatis porin A (MspA)3 with phi29 DNA polymerase (DNAP)4, which controls the rate of DNA translocation through the pore. As phi29 DNAP synthesizes DNA and functions like a motor to pull a single-stranded template through MspA, we observe well-resolved and reproducible ionic current levels with median durations of ~28 ms and ionic current differences of up to 40 pA. Using six different DNA sequences with readable regions 42–53 nucleotides long, we record current traces that map to the known DNA sequences. With single-nucleotide resolution and DNA translocation control, this system integrates solutions to two long-standing hurdles to nanopore sequencing
文献链接:https://www.nature.com/nbt/journal/vaop/ncurrent/full/nbt.2171.html