罗丽娟1 ,2 ,施季森1 3
(1. 南京林业大学,江苏 南京 210037 ;2. 华南热带农业大学,海南 儋州 571737)
摘 要:在分子生物学研究中,基因克隆和分子杂交的探针制备等操作常需分离与已知DNA序列邻近的未知序列,热不对称交错PCR(简称TAIL-PCR) 反应技术能够较好地解决上述难题。该技术通过3 个嵌套的特异性引物分别和简并引物组合进行连续的PCR 循环,利用不同的退火温度选择性地扩增目标片段,所获得的片段可以直接用做探针标记和测序模板。TAIL-PCR 技术简单易行,反应高效灵敏,产物的特异性高,重复性好,能够在较短的时间内获得目标片段,已经成为分子生物学研究中的一种实用技术。笔者综述了 TAIL-PCR 反应的原理、引物设计、反应条件设置及产物分析。
关键词:热不对称交错PCR ;特异性引物;简并引物;侧翼序列分离
TAIL-PCR:A Simple and Eff icient Method to Isolate DNA Segments Adjacent to Known Sequence
LUO Li-juan1 ,2 ,SHI J i-sen1 3
(1. Key Laboratory of Forest Genetics and Gene Engineering ,Nanjing Forestry University ,Nanjing 210037 ,China ;
2. South China University of Tropical Agriculture ,Danzhou 571737 ,China)
Abstract :Isolation of DNA f ragment adjacent to a known sequence is a tedious task in genome-related research. Thermal asymmet ric interlaced PCR ( TAIL-PCR) is an efficient technique for isolation of target DNA segment s flanking known sequences. It was utilized three nested sequence-specific primers with a shorter arbit rary degenerated(AD) primer respectively. Specific product s can be amplified through continuous cycles of amplification at different annealing temperature. The amplified product s are suitable for either hybridization probes or sequencing templates. TAIL-PCR technique is very simple and highly efficient . The specific product s can be obtained again in the same PCR mixture and the same PCR condition. All the TAIL-PCR reaction need only a few time as in one day. It has become a powerful tool in a genomic analysis.This article present s the principle and protocol of TAIL-PCR. Methods for primers design and product s selection are also discussed.
Key words :TAIL-PCR ;Special prime ;Arbit rary degenerate prime ;Flanking sequence isolation
热不对称交错PCR( Thermal Asymmet ric Interlaced PCR ,简称TAIL-PCR) 是一种用来分离与已知序列邻近的未知DNA 序列的分子生物学技术。该技术由Liu 和Whitter 首先研究并报道[1 ] 。在分子生物学研究领域中,利用该技术分离出的DNA 序列可以用于图位克隆、遗传图谱绘制的探针,也可以直接测序。目前已成功地从P1 、YAC 和BAC 克隆中分离获得插入末端的DNA 序列和拟南芥( A rabidopsis thaliana L. ) 的T-DNA 侧翼序列[1~3 ] 。此外,经过改良的TAIL-PCR 技术能够快速克隆Pal 及Pgi 基因的启动子序列和野油菜黄单胞菌群体感应信号基因[4 ,5 ] 。该技术的问世,使分子生物学研究工作者能够简易而有效地从已知序列中分离到其邻近的未知侧翼序列,解决有关基因操作的一系列难题。现对此技术的原理、技术要点和难点作一综述。
1 TAIL-PCR 技术的原理
由一个特异性引物和一个简并引物相组合构成的PCR 反应叫做“半特异性PCR”[6 ,7 ] 。这种反应会产生3 种不同类型的产物: (1) 由特异性引物和简并引物扩增出的产物; (2) 由同一特异性引物扩增出的产物; (3) 由同一简并引物扩增出的产物。在TAIL-PCR 反应中,后两种非目标产物可以通过以嵌套的特异性引物进行的后续反应来消除。TAIL-PCR 技术的基本原理是利用目标序列旁的已知序列设计3 个嵌套的特异性引物( special prime ,简称sp1 ,sp2 ,sp3 ,约20 bp) ,用它们分别和1个具有低Tm 值的短的(14 bp) 随机简并引物(Arbit rary degenerate prime 简称AD) 相组合,以基因组DNA 作为模板,根据引物的长短和特异性的差异设计不对称的温度循环,通过分级反应来扩增特异引物。反应流程如图1 所示。
图1 TAIL-PCR 反应流程图
Fig. 1 The protocol of thermal asymmetric interlaced PCR
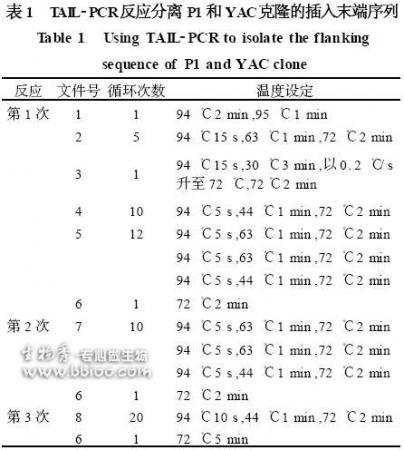
TAIL-PCR 一般分为3 次反应(图1) 。第1 次PCR 反应由5 次高特异性反应、1 次低特异性反应、10 次较低特异性反应和12 次热不对称的超级循环构成。通过5 次高特异性的反应,使sp1 与已知的序列(载体或T2DNA 等) 退火并延伸,提高目标序列的浓度。1 次低特异性的反应使简并引物结合到较多的目标序列上,10 次较低特异性的反应使两种引物均能与模板退火,随后进行12 次TAIL 循环。经过上述反应得到了不同浓度的3 种类型的产物:特异性产物( Ⅰ型) 和非特异性产物( Ⅱ型和Ⅲ型) 。第2 次反应则将第一级反应的产物稀释1000 倍作为模板,通过10 次热不对称的超级循环,使特异性的产物被选择性地扩增,而非特异性的产物被压制到极低的含量。第3 次反应又将第2 次反应的产物稀释1 000 倍作为模板,一般设置为普通的PCR 反应或热不对称的超级循环。通过上述3 次PCR 反应可获得与已知序列邻近的目标序列。
2 TAIL-PCR 的反应条件和引物设计
2. 1 TAIL-PCR的反应条件
TAIL-PCR 反应的条件在不同的实验中稍有变化。在表1 所示的条件下对P1 和YAC 克隆插入末端序列的分析中获得了满意的结果[1 ] 。这种反应条件亦是一个规范的实验设置,可以满足大多数实验的需要。
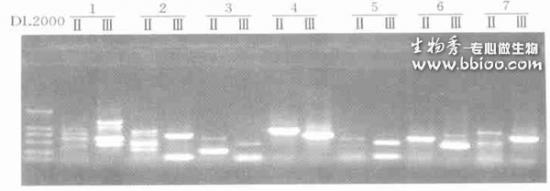
笔者用TAIL- PCR 方法研究T2DNA 标记的水稻( Oryz a sativa L. cv. Nipponbare) 突变体,用表2 所示的反应条件有效地分离到了T2DNA 侧翼序列(图2) 。图2 中第3 次反应产物( Ⅲ) 中的明亮条带即为扩增到的目标序列。
>
2. 2 TAIL-PCR的引物设计
根据载体的已知序列设计3 个与其边界距离不等的嵌套的特异性引物,特异性引物的长度约20 bp , Tm 一般为58~68 ℃。再按照物种普遍存在的蛋白质的保守氨基酸序列设计一系列简并引物,简并引物相对较短,长度为14 bp , Tm 为30~48 ℃。为了增加简并引物与目标序列间退火的可能性,除了3′端的3 个碱基以外,其它位置的碱基包含简并核苷酸。特异性引物和简并引物的选择直接影响扩增的效果。如果不能获得满意的扩增结果,则应该在预备实验中重新设计特异性引物或者换用其它的简并引物。在TAIL-PCR 反应中,高特异性循环的退火温度设为65 ℃左右,较低特异性循环的退火温度设为44 ℃,低特异性循环的退火温度设为25~30 ℃。反应体系中,特异性引物的浓度与普通的PCR 相同,简并引物的浓度要高,一般为2. 5~5. 0μmol/ L ,以满足引物的结合效率。
3 TAIL-PCR 产物分析
在多数情况下,第1 次反应有较多的非特异性产物。由sp1 和AD 为引物扩增出的特异性片段在第1 次反应中由于量少而不一定能显现出来。当用sp2 替代sp1 进行第2 次反应后,一些非特异性条带消失,特异性的产物被选择性地扩增而显现。欲增加产物的特异性,可以再把第3 次反应设置为TAIL 循环。由于特异性引物在第1 次反应中一般不能扩增到可被检测的水平,因此可以省去第1 次反应产物的琼脂糖凝胶电泳分析,仅仅检测第2 次和第3 次反应的产物来获得特异性的目标片段。取第2 次反应产物和第3 次反应产物成对点样。由于特异引物是嵌套的,因此特异性扩增的特点是第3 次反应产物小于第2 次反应产物。不同的AD 引物的扩增效率不同。有效的TAIL-PCR 扩增出的目标片段的大小应该在250 bp~2 kb。由于AD 引物在特异性引物的下游有多个退火位点,同一目标序列有时会产生一个以上不同长度的特异性片段[1 ] 。
4 TAIL-PCR 的优点以及技术难点
常规的PCR 只能扩增两引物之间的DNA 区段,难以对已知DNA 序列侧翼的未知序列进行扩增。Reverse PCR、Hemispecific、one2sided PCR 等方法可以部分地克服上述缺点,但在PCR 反应前必须作酶切、连接、加尾等一系列繁琐的操作。TAIL-PCR 技术能够快速地分离到目标序列,对基因克隆研究具有重要的意义。TAIL-PCR 技术有以下优点: (1) 简单。只要设计好引物,即可以用基因组DNA 作模板直接筛选到目标序列。节省了PCR 反应前后的许多费时、费力的操作程序。只需几纳克的DNA 即可作为模板,对其纯度的要求也不很高。(2) 特异性高。用短的简并引物和长的特异性嵌套引物相组合,通过不对称的温度循环和分级反应,使反应体系有利于特异引物的扩增,最终的扩增产物中目的片段占绝对优势,反应产物可以直接用做探针标记和测序模板。(3) 高效灵敏。使用任何一个AD 引物,在60 %~80 %的反应中能够产生特异性产物。运用不同的AD 引物就能够有效地扩增到目标片段。(4)快速。整个TAIL-PCR 反应循环能够在1 d 内完成,可以快速地获得目标片段。(5) 不涉及连接反应,反应产物准确可靠,重复性好。
TAIL-PCR 反应需要较多的引物组合。此外,由于AD 引物存在有限的结合位点,对于个别的侧翼序列,即使使用不同的简并引物也难以扩增到阳性结果。整个TAIL-PCR 需要一系列连续的反应,反应条件的设置要求比较精细。
TAIL-PCR 技术能够分离获得克隆载体上的DNA 序列,也能够用于基因组小的物种如拟南芥[3 ,8 ] 、水稻[9 ,10 ]和基因组大的物种,如小麦[1 ]以及哺乳动物[11 ]的已知序列两侧翼的DNA 序列的分离。上述优点使TAIL-PCR 技术成为分子生物学研究中的一种强大工具。
[ 参考文献]
[ 1 ] Liu Yao-Guang ,Robert F Whitter. Thermal asymmetric interlaced PCR :automatable amplification and sequencing of insert end fragment from P1 and YAC clones for chromosome walking[J ] . Genomics ,1995 ,25 :674 - 681.
[ 2 ] Liu Yao2Guang ,Huang Ning. Efficient amplification of insert end sequences from bacterial artificial chromosome clones by thermal asym-metric interlaced PCR[J ] . Plant Molecular Biology Reporter ,1998 ,16 (2) :175 - 181.
[ 3 ] Liu Yao2Guang ,Norihiro Mitsukawa ,Teruko Oosumi ,Robert F Wittier. Efficient isolation and mapping of A rabi dopsis thaliana T-DNA
insert junctions by thermal asymmetric interlaced PCR[J ] . The Plant Journal ,1995 ,8 (3) :457 - 463.
[ 4 ] Terauchi R , Kahl G. Rapid isolation of promoter sequences by TAIL-PCR : the 5′2 flanking regions of Pal and Pgi genes from Yams(Dioscorea) [J ] . Mol Gen Genet ,2000 ,263 :554 - 560.
[ 5 ] 应 革,武 威,何朝族. TAIL-PCR 方法快速分离XCC 致病相关基因序列[J ] . 生物工程学报,2002 ,18 (2) :182 - 186.
[ 6 ] Forman M A ,DUSH M K,Martin G R. Rapid production of full2length cDNA rare transcripts :amplification using a single gene2specific oligonucletide prime[J ] . Proc Natl Acad Sci USA ,1988 ,85 :8 998 - 9 002.
[ 7 ] Ohara O ,Dorit R L ,Gilbert . One2sided polymerase chain reaction :the amplification of cDNA[J ] . Proc Natl Acad Sci USA ,1989 ,86 :5 673 - 5 677.
[ 8 ] Ueli Grossniklaus ,Jean2Philippe ,Vielle2Calzada. Maternal control of embrogenesis by M EDEA ,a polycomb group gene in A rabi dopsis[J ] .Science ,1998 ,280 :446 - 450.
[ 9 ] Chin Hang Geong ,Chin Hang Gyeong ,Mi Sook Choe , et al . Molecular analysis of rice plants harboring an Ac/ Ds transposable element2mediatd gene trapping system[J ] . The Plant Journal ,1999 ,9 (5) :615 - 623.
[ 10 ] 方 进,翟文学,王文明,等. 转基因水稻T2DNA 侧翼序列的扩增与分析[J ] . 遗传学报,2001 , 28 (4) :345 - 351.
[ 11 ] Hahn S A ,Schutte M ,Hooue A , et al . A candidate tumer2suppressor gene at human2chromosome[J ] . Science ,1996 , 271 :350 - 353.