
近年来基因组测序技术发展迅猛,为各种疾病的研究打开了一扇新窗口,比如癌症研究领域,就在新型测序技术的基础上获得了不少重要的成果,近期Cell,Nature Genetics等顶级刊物也发表了这方面的成果。
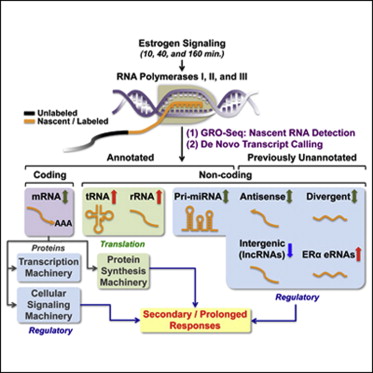
A Rapid, Extensive, and Transient Transcriptional Response to Estrogen Signaling in Breast Cancer Cells
来自美国康奈尔大学,德州大学西南医学中心等处的研究人员利用一种新型的测序技术完成了目前最全面的雌激素主要瞬即效应的检测,首次表明雌激素对乳腺癌细胞的作用是如此直接和广泛,这对于乳腺癌的基础研究及治疗都具有重要的意义。这一研究成果公布在Cell杂志上。
科学家们早些年就已知道雌激素在乳腺癌细胞中起着重要的作用,约2/3的乳腺癌细胞都有雌激素受体表达,这些受体可将雌激素作为有丝分裂信号产生响应。雌激素是乳腺癌生长的首要驱动力,雌激素受体可以成为激素抑制治疗药物(如他莫西芬)的作用靶点以及预后判断指标,因此这方面的研究也受到了许多大型实验室的关注。
在这篇文章中,研究人员利用一项叫GRO-seq(Global Run-On Sequencing)测序技术,并通过一种新的生物信息学方法来分析数据,发现雌激素信号能快速,有力,并以一种出乎预料的短暂方式直接调节很大部分的转录谱。除编码蛋白的基因外,雌激素还调控了全部三种RNA聚合酶的分布和活性,事实上还包含迄今为止所有描述过的每一类非编码RNA,研究人员还首次发现数千种以往未注明或未检测到的由雌激素调节的基因间转录,为进一步研究开辟了新的方向。这为引发细胞分裂的雌激素依赖作用提供了一个有关的更广的基因调解谱图。
这种GRO-seq技术可以在整个基因组范围上绘制参与转录的RNA聚合酶的位置、数量和定位。通过这项技术,研究人员可以在短时间内对雌激素在分子水平对细胞生长调节作用进行观察。在此之前,研究人员需要基于稳态基因表达方式或需耗费数小时或数天的静态成像技术,对根据DNA序列制备RNA拷贝的基因转录过程进行干扰以便进行观察。现在,通过GRO-seq技术,研究人员可以观测到基因组水平的进行的转录,这是一种对所有参与DNA聚合酶位置和定向的高分辨率虚拟图谱。
除此之外,这项研究还采用了一种新型生物信息学方法来分析数据,这种方法可以直接运用GRO-seq的数据来发现转录物。这样研究人员还发现了许多未曾被发现的雌激素调控的基因间转录物,其中有很多与雌激素受体的结合位点相邻。因此可以说这是目前进行的最全面雌激素主要、瞬即效应的测量,这为理解其他系统中信号依赖的快速转录进行提供了资源。
Genomic sequencing of colorectal adenocarcinomas identifies a recurrent VTI1A-TCF7L2 fusion
来自美国Dana-Farber癌症研究所(Dana-Farber Cancer Institute)、哈佛医学院、布洛德研究所(Broad Institute)、以色列魏兹曼科学等处的研究人员对大肠癌样品展开了全基因组测序分析,并从中发现了与大肠癌发病相关的一个新的基因融合。新研究发现为进一步进入了解大肠癌的发病演化机制提供了新线索。
研究人员利用Illumina GAII的测序平台对9例大肠癌肿瘤组织及匹配的正常组织进行了全基因组测序分析,其测序深度分别达到30.7倍和31.9倍。根据基因组分析的结果,研究人员发现了137,968个潜在的体细胞突变,其中的712个有可能导致了蛋白质编码序列碱基错义替换(non-synonymous substitution)、插入或缺失。研究人员利用质谱基因分型法对521个缺失突变进行了检测,证实其中的439个是真正的体细胞突变。
基于基因组分析的结果,研究人员推测出每个大肠癌基因组大约每10-6个序列包含5个突变,其中基因间序列大约为每10-6个序列包含6.7个突变,内含子序列大约为每10-6个序列包含4.8个突变,外显子序列大约为每10-6个序列包含4.2个突变。此外,研究人员还粗略推算出在外显子序列中大约每10-6个序列就包含3.1个错义突变。研究人员指出与过去的研究相一致的是,他们发现突变主要集中在基因组的CpG位点。
在随后研究人员在对另一组的97个结肠癌肿瘤样品进行筛查时,在3例癌症样本中发现了一个新的基因融合VTI1A -TCF7L2。
新研究第一次揭示了与大肠癌发病相关的一个基因融合,这一研究发现或将帮助医生更有效地开展临床治疗,并推动研究人员开发出新治疗药物及大肠癌癌诊断新工具。
相关英文原文摘要:
A Rapid, Extensive, and Transient Transcriptional Response to Estrogen Signaling in Breast Cancer Cells
We report the immediate effects of estrogen signaling on the transcriptome of breast cancer cells using global run-on and sequencing (GRO-seq). The data were analyzed using a new bioinformatic approach that allowed us to identify transcripts directly from the GRO-seq data. We found that estrogen signaling directly regulates a strikingly large fraction of the transcriptome in a rapid, robust, and unexpectedly transient manner. In addition to protein-coding genes, estrogen regulates the distribution and activity of all three RNA polymerases and virtually every class of noncoding RNA that has been described to date. We also identified a large number of previously undetected estrogen-regulated intergenic transcripts, many of which are found proximal to estrogen receptor binding sites. Collectively, our results provide the most comprehensive measurement of the primary and immediate estrogen effects to date and a resource for understanding rapid signal-dependent transcription in other systems.
Genomic sequencing of colorectal adenocarcinomas identifies a recurrent VTI1A-TCF7L2 fusion
Prior studies have identified recurrent oncogenic mutations in colorectal adenocarcinoma1 and have surveyed exons of protein-coding genes for mutations in 11 affected individuals2, 3. Here we report whole-genome sequencing from nine individuals with colorectal cancer, including primary colorectal tumors and matched adjacent non-tumor tissues, at an average of 30.7× and 31.9× coverage, respectively. We identify an average of 75 somatic rearrangements per tumor, including complex networks of translocations between pairs of chromosomes. Eleven rearrangements encode predicted in-frame fusion proteins, including a fusion of VTI1A and TCF7L2 found in 3 out of 97 colorectal cancers. Although TCF7L2 encodes TCF4, which cooperates with β-catenin4 in colorectal carcinogenesis5, 6, the fusion lacks the TCF4 β-catenin–binding domain. We found a colorectal carcinoma cell line harboring the fusion gene to be dependent on VTI1A-TCF7L2 for anchorage-independent growth using RNA interference-mediated knockdown. This study shows previously unidentified levels of genomic rearrangements in colorectal carcinoma that can lead to essential gene fusions and other oncogenic events.
英文原文链接:https://www.nature.com/ng/journal/v43/n10/full/ng.936.html
https://www.sciencedirect.com/science/article/pii/S009286741100376X







