本文转载自“西湖欧米”公众号。
导语:COVID-19仍在全球肆虐,且似乎不会从地球消失。在这篇评述中,Lu等人讨论了多组学技术在揭示COVID-19发病、进展和传播背后的分子机制中所起的作用,并讨论了标准化的缺失如何限制了它们在临床中的应用。
2022年3月15日,西湖大学生命科学与生物医学实验室团队在Cell Reports Medicine发表了题为“Multi-omics in COVID-19:Seeing the unseen but overlooked in the clinic”的文章(图1)[1]。

图1 研究成果(图源:Cell Reports Medicine)
截至2022年2月12日,200多个国家和地区报告了超过4.05亿例COVID-19确诊病例及580万例死亡病例。虽然疫苗接种并没能阻止SARS-CoV-2的传播,但却降低了成人和儿童感染后发展成重症或死亡的风险。多组学技术研究揭示了病原体的分子特征与结构,以及宿主对病毒感染和疫苗接种后反应的潜在分子机制(图2和图3)。
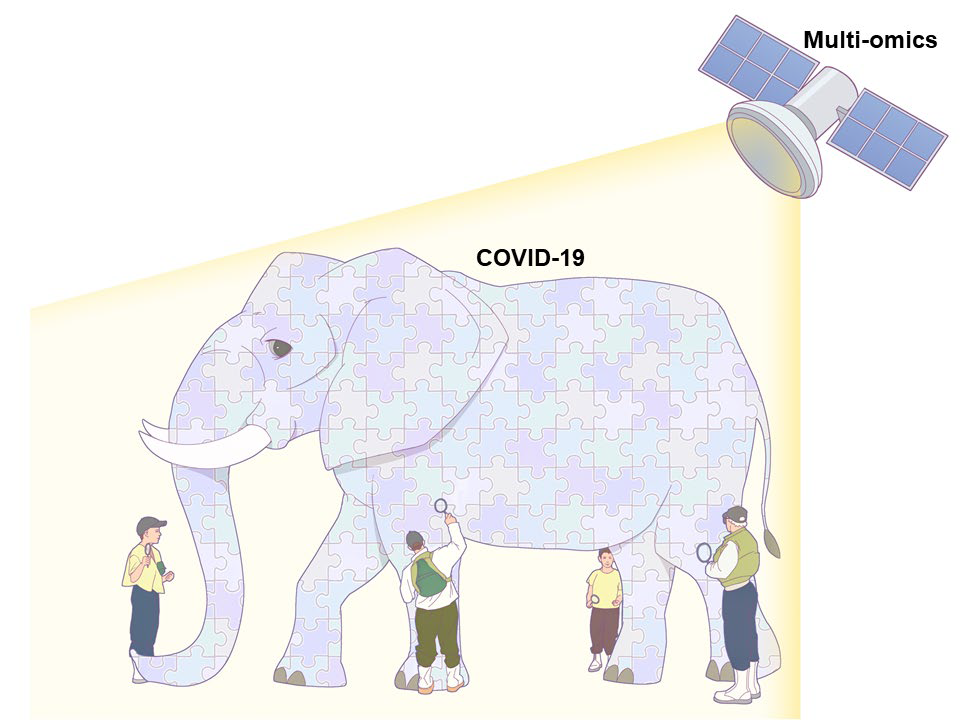
图2 多组学能够系统地表征COVID-19中的分子调节(图源:Cell Reports Medicine)
注:大象指的是COVID-19患者,而放大镜指用于检查有限分子数量的传统技术。多组学技术,就像卫星一样,可以全面分析分子调控机制,其中大部分是放大镜看不到的东西。
一、病原体
多组学技术极大加深了我们对SARS-CoV-2以及宿主反应的理解,这些技术包括但不限于第二代测序(Next-Generation Sequencing,NGS)、蛋白质组学和代谢组学(图 3)。事实上,新冠大流行病原体的确定还需要归功于基因组学。基于这个结果,人们开发了PCR试剂盒,并将之迅速应用于临床,取代了原始的体温检测法,有效提高了COVID-19诊断的准确性。
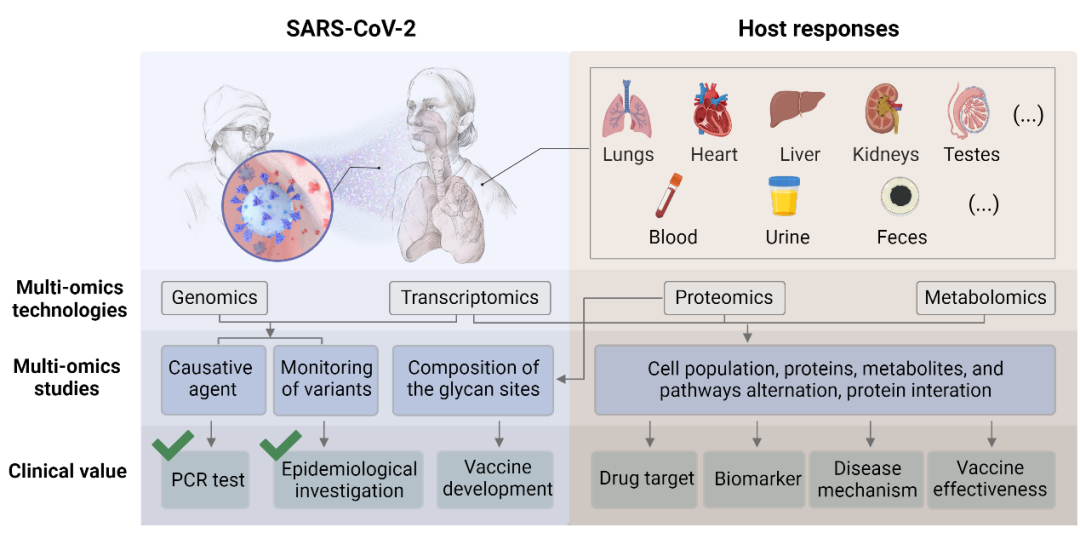
图3 关于SARS-CoV-2和宿主反应的多组学研究(图源:Cell Reports Medicine)
注:改编自由BioRender.com(2022年)撰写的“SARS-CoV-2关注变体的潜在传播机制”;来源于https://app.biorender.com/biorender-templates;病毒的基因组和蛋白质组研究让人们快速建立了用于COVID-19诊断的PCR检测方法。基因组的监测使我们能够对病毒的进化和传播进行流行病学调查。目前已经根序列和表面蛋白设计了多种疫苗。大多数多组学分析都集中在宿主对感染或疫苗接种的反应上,蛋白质组学和代谢组学是这方面最重要的主力军。
SARS-CoV-2的突变会导致其致病性、传染性、传播性或抗原性的变化。例如,研究发现E484K是一种免疫逃逸突变,可以减少抗体介导的中和作用。NGS技术的迅速应用不仅有助于Delta和Omicron等突变毒株的发现,而且有助于跟踪调查它们在不同地区的传播情况[1]。在津巴布韦对SARS-CoV-2变异毒株流行病学调查的一项研究中发现,60%的病例是输入性的,这表明人口流动是其传播的关键因素,进一步支持了检疫和限制人口流动的重要性[4]。基于质谱(MS)的对新冠病毒刺突(S)糖蛋白质的研究也有助于疫苗的研发。病毒的S蛋白会与宿主细胞中的ACE2蛋白产生相互作用,利用这一点,抗体通过靶向作用于S蛋白来中和病毒。因此,S蛋白的结构,包括其糖基化位点,对疫苗的设计十分关键。Watanabe等人表达了重组刺突蛋白三聚体,并通过质谱确定了22个糖基化位点的糖基组成(例如,N234和N709位点上糖基主要属于寡甘露糖型)[4]。这个研究不仅深化了我们对S蛋白结构的了解,而且还为评估疫苗和治疗性抗体开发中的免疫原质量提供了基准。
二、宿主对病原体的反应
虽然多组学帮助我们在了解病原体的性质和结构上取得了一定的进展,但新冠病毒感染患者的症状,如哪些患者会发展成重症,仍然难以预测。如果有一种精确而实用的方法来根据疾病严重程度对患者进行分类,并确定那些能在SARS-CoV-2感染中幸存下来的大多数患者,那么许多对全球大流行的担忧就可以得到缓解。此外,不同人群对新出现的突变毒株的免疫反应是否存在差异?不同的疫苗和接种次数是如何影响人体对SARS-CoV-2的免疫力的?要解决这些问题及许多其他关键问题,我们需要全面了解宿主对这种病原体及其疫苗的反应。大多数多组学研究能够基于多种器官和临床标本,探究宿主对病原体的成千上万种分子变化,这是传统的方法难以实现的。
COVID-19宿主反应研究最初仅限于观察如发烧和咳嗽等的临床症状,进一步延伸到观察胸部CT及如CRP和SAA1等的循环蛋白生物标志物指标的变化。这些标志物已用于其他传染病的观察,基于这些经验,它们也被用于监测COVID-19的临床进展。多组学研究监测了人体内大量由于感染新冠发生改变的分子,极大地拓展潜在的标志物种类,这些发现通常难以用传统的分析方法检测到(图3)。例如,基于COVID-19患者血清的蛋白质组学和代谢组学研究已有效地应用于测量约2000个血液中的分子,其中93种蛋白质和204种代谢物在重症患者中表现出显著的特异性失调。该研究还强调了血小板脱颗粒、巨噬细胞和补体系统的关键作用。大多数关键分子变化已在多个国家/地区的其他新冠多组学研究中得到证实,验证了蛋白质组学和代谢组学技术的可靠性以及他们在临床决策中的潜在价值[4]。
值得一提的是,尿液作为一种非侵入性的临床生物标本,其中包含的蛋白质和小分子代谢物也可以很容易地被质谱检测到。研究人员在尿液中检测到了近4000种蛋白质,而由于血清中高丰度蛋白质的存在,使用相同的质谱方法仅能检测到约1500种蛋白质。值得注意的是,可在血清中检测到的蛋白有80%可在尿液中检测到,而在尿液中检测到的蛋白仅有31%可以在血清中检测到[6]。
为什么我们需要检测这些分子呢?这些发现对抗疫有何帮助?多项组学研究表明,在经过合理设计的组学实验中,机器学习可以筛查用于分类和监测疾病进展的生物标志物。例如,基于血清中表达的22种蛋白质和7种代谢物的随机森林模型可以可靠地指示 COVID-19的严重程度,并在一定程度上预测疾病预后。4同样,基于尿液蛋白的模型也可用于监测COVID-19疾病进展[6]。
除了血液中的循环分子外,其他器官中的分子变化对于揭示宿主反应也至关重要。病理切片检查在宏观和微观层面为诊断提供了丰富的信息,被广泛认可为疾病诊断的“金标准”。然而,在新冠患者的多器官病理切片中观察到的改变与非典型性肺炎(SARS)和中东呼吸综合征(MERS)患者中呈现的的组织病理学区别并不明显,而是高度相似。基于质谱的蛋白质组学现在能够在多器官尸检样品中表征超过10000种蛋白质,并发现了参与凝血,血管生成,纤维化和脂肪酸代谢等过程的多种失调蛋白质[7]。
如果没有这样的高深度的蛋白质组学技术,而使用基于假设的研究方法来探索新冠感染后人体的变化,那么目前已知的几个重要发现,如肺部组织蛋白酶L(Cathepsin L)的上调与COVID-19死亡率之间存在潜在联系或INSL3蛋白减少与睾丸中间质细胞受损之间的关联,将耗费无法估量的精力与时间[8]。蛋白质组学使我们能够“看到”病理表现背后的分子变化。患者的组织标本,特别是肺组织,可能具有传染性,在分析之前必须进行灭活,这通常通过福尔马林固定来实现,而其通常会导致mRNA降解,使得基于福尔马林固定的样本的转录组学分析存在挑战性。相比之下,蛋白可以被有效地从福尔马林固定的组织中提取,然后进行质谱分析[9]。
组学技术还能够发现病毒的靶细胞,这是传统技术难以实现的。鼻咽拭子的单细胞RNA 测序(single-cell RNA sequencing,scRNA-seq)显示,在COVID-19感染期间,咽部的纤毛细胞(ciliated cell)减少,同时分泌细胞(secretory cell)、次胞质体 (deuterosomal cell)扩增和巨噬细胞(macrophages)增加。没有其他技术可以替代单细胞RNA测序直接从临床标本中获得这样的结果[10]。不仅如此,scRNA-seq也可以用于解码同种细胞的多种亚细胞群。例如,淋巴细胞减少症(血液中循环淋巴细胞的显著减少)是COVID-19患者的常见症状。各种淋巴细胞在抗病毒免疫中发挥着不同的作用,基于这些淋巴细胞的scRNA-seq研究发现了三种不同淋巴细胞亚细胞群响应模式与患者的预后有关[11]。
三、对疫苗的反应
多组学研究还可能为疫苗的使用提供指导意见,尽管这些数据目前尚不充分。全球已接种超过100亿剂疫苗,包括mRNA疫苗、灭活病毒疫苗和基于腺病毒的疫苗等。虽然所有疫苗都对预防SARS-CoV-2感染起到了一定的保护作用,但mRNA疫苗被证实比其他疫苗带来了更高的中和抗体滴度。然而,这种保护作用不仅通过中和抗体起作用,而且还由其他免疫效应机制介导,包括T细胞和先天免疫细胞[12]。例如,在没有检测到中和抗体的情况下,即使只接种一剂,mRNA疫苗对预防感染也达到了80%的有效性。11此外,高达6%的被接种人在第二次注射后血清中仍然检测不到中和抗体。
据美国疾控中心报道,截至2022年2月12日,超过0.004%的被接种人出现了严重的副作用,包括过敏反应、血栓形成伴血小板减少综合征、吉兰-巴雷综合征甚至死亡[12]。中和抗体的浓度随时间推移而下降,突破性感染时有发生,尤其是当面对新兴的免疫逃逸突变毒株如Omicron时。迄今为止,针对特定突变毒株的定制疫苗尚未出现,尽管其在预防COVID-19方面具有不可否认的重要性[13]。解决疫苗的保护性免疫不确定性的一项有争议但务实的政策是强制加强疫苗的接种。

疫苗接种的这些局限性,是目前多个国家数亿被接种人次所遇到的普遍问题。理论上,通过实时监测血清抗体含量变化可以为人群的流行病学追踪提供信息,更重要的是,可能为指导疫苗剂量和接种间隔提供有用的信息。然而,即使是中和抗体的检测也没有被广泛用于指导临床决策,这可能是因为大规模血液采样的可行性有限,并且缺乏通用的标准流程。监测尿蛋白的变化可能是评估对病毒免疫力的更实际的手段,但是,目前也还没有相关的数据公布。
四、为什么临床实践很大程度上忽视了蛋白质组学和代谢组学?
虽然已经发表的与COVID-19相关的组学论文已有近10000篇(截至2022年2月12日的PubMed搜索),但除了PCR和病毒的基因组分析外,大部分组学研究的结果尚未成功进入COVID-19的临床应用。一个可能的原因是,人们的认知中多组学技术还不成熟(主要指蛋白质组学和代谢组学),重复性和可靠性不高,无法提供临床上可靠的结果。
然而,最近世界各地的多个实验室发表的蛋白质组学数据在失调的分子的种类和变化趋势上显示出惊人的一致。相比之下,众所周知的是假阴性在SARS-CoV-2的PCR检测中很常见。最近一项针对95919名患者的研究报告显示PCR检测新冠病毒的假阴性率 (False-Nnegative Rate,FNR)和敏感性分别为9.3%(95% CI 1.5–17.0%)和 90.7%(95% CI 82.6–98.9%)。有些人可能会争辩说,蛋白质太不稳定了,在实际应用中难以成为稳定可信的生物标志物[14]。然而,研究表明,通过质谱测量的蛋白质比 NGS测量的mRNA更稳定[15]。

那是因为蛋白质和代谢物的测量在技术和分析上太具有挑战性,而且成本太高吗?确实,蛋白质组学和代谢组学数据的获取需要许多步骤,依赖于特殊的专业知识和昂贵的仪器。例如,蛋白质组学的数据采集依赖于高分辨率质谱仪。然而,更复杂和昂贵的技术如正电子发射断层扫描(Positron Emission Tomography,PET)等已经在临床上广泛使用,所以这些也不应该是不可逾越的障碍。此外,通过质谱法测量一种蛋白质的成本已从2006年的约3美元降至2020年的不到0.1美元[16]。
蛋白质组学和代谢组学临床应用的主要障碍可能是缺乏标准化。由于蛋白质组学领域正在快速发展,不同实验室几乎不会使用完全相同的标准操作流程(Standard Operating Protocols,SOP);而代谢组学的情况更加令人困惑,出于商业考虑,即使是在不同实验室中生成的原始数据文件也很少得到共享。尽管代谢组学的商业化先于蛋白质组学的商业化,但大多数代谢组学平台并不公开其开发的资源(例如,用于分子测量的化合物库)和技术,甚至在科学交流中也不共享其原始数据。想要实现必要的透明度和标准化,扭转这种令人气馁的局面绝非易事。相比之下,蛋白质组学界的状况更加科学透明、心态更加开放共享。我们相信,在不久的将来,通过蛋白质组学科学家、临床从业者和其他利益相关者的共同努力,可以制定出几个能够获得国际共识的用于临床的蛋白质组学SOP。
五、其他挑战
整合对相同样本使用不同组学方法测量的数据对多组学的临床应用也是一大挑战。基因组数据大多是定性的,而转录组、蛋白组和代谢组的数据大多是定量的。因此,人们很难通过算法直接分析这复杂多样的数据。此外,与在相同个体的不同组织之间保持一致的基因组不同,蛋白质组和代谢组具有组织特异性和环境依赖性,能够反映生理和病理上的复杂的实时状态,其时空异质性也为数据采集和分析带来巨大的挑战。
除了这些技术限制之外,在大流行期间收集潜在的传染性样本还带来了额外的挑战。COVID-19研究中使用的最常见样本是血液或支气管肺泡灌洗液样本,而尿液和粪便等非侵入性样本的数据也已逐渐被证明能够有效反映宿主反应。然而,医院缺乏用于安全收集和有效预处理这些样本的SOP以进行多组学研究,这可能成为队列内和不同队列之间偏倚的来源之一。考虑到生物安全的问题,对COVID-19样本的获取有诸多限制,但这也限制了对这些珍贵样本的进行全面深入研究的可能,从而限制了人们对COVID-19 的探索来发展有效防疫措施。如果能够建立适当的处理和获取这些潜在传染性标本且技术上可行的SOP,我们对COVID-19的研究将会更快地发展和深入。COVID-19不太可能消失,这也不会是最后一次大流行病。因此,人类绝不能自满于目前所取得的成就。国际社会现在必须采取行动,为未来做好更充分的准备。
排版|乔维钧
参考资料:
[1]Tian Lu, Yingrui Wang, Tiannan Guo, Multi-omics in COVID-19: Seeing the unseen but overlooked in the clinic, Cell Reports Medicine, 2022 Mar;3(3):2666-3791.doi:10.1016/j.xcrm.2022.100580.
[2]Harvey WT, Carabelli AM, Jackson B, et al. COVID-19 Genomics UK (COG-UK) Consortium, Peacock SJ, Robertson DL. SARS-CoV-2 variants, spike mutations and immune escape. Nat Rev Microbiol. 2021 Jul;19(7):409-424. doi: 10.1038/s41579-021-00573-0. Epub 2021 Jun 1. PMID: 34075212; PMCID: PMC8167834.
[3]Mashe T, Takawira FT, de Oliveira Martins L, et al. COVID-19 Genomics UK Consortium; SARS-CoV-2 Research Group. Genomic epidemiology and the role of international and regional travel in the SARS-CoV-2 epidemic in Zimbabwe: a retrospective study of routinely collected surveillance data. Lancet Glob Health. 2021 Dec;9(12):e1658-e1666. doi: 10.1016/S2214-109X(21)00434-4. Epub 2021 Oct 22. PMID: 34695371; PMCID: PMC8536247.
[4]Watanabe, Y., Allen, J.D., Wrapp, D., et al. Site-specific glycan analysis of the SARS-CoV-2 spike. Science 369, 330-333 (2020).
[5]Shen B, Yi X, Sun Y, et al. Proteomic and Metabolomic Characterization of COVID-19 Patient Sera. Cell. 2020 Jul 9;182(1):59-72.e15. doi: 10.1016/j.cell.2020.05.032. Epub 2020 May 28. PMID: 32492406; PMCID: PMC7254001.
[6]Bi X, Liu W, Ding X, et al. Proteomic and metabolomic profiling of urine uncovers immune responses in patients with COVID-19. Cell Rep. 2022 Jan 18;38(3):110271. doi: 10.1016/j.celrep.2021.110271. Epub 2021 Dec 28. PMID: 35026155; PMCID: PMC8712267.
[7]Deshmukh, V., Motwani, R., Kumar, A., et al. Histopathological observations in COVID-19: a systematic review. J Clin Pathol 74, 76-83 (2021).
[8]Nie X, Qian L, Sun R, et al. Multi-organ proteomic landscape of COVID-19 autopsies. Cell. 2021 Feb 4;184(3):775-791.e14. doi: 10.1016/j.cell.2021.01.004. Epub 2021 Jan 9. PMID: 33503446; PMCID: PMC7794601.
[9]Zhu Y, Weiss T, Zhang Q, et al. High-throughput proteomic analysis of FFPE tissue samples facilitates tumor stratification. Mol Oncol. 2019 Nov;13(11):2305-2328. doi: 10.1002/1878-0261.12570. Epub 2019 Sep 18. PMID: 31495056; PMCID: PMC6822243.
[10]Ziegler CGK, Miao VN, Owings AH, et al. Impaired local intrinsic immunity to SARS-CoV-2 infection in severe COVID-19. Cell. 2021 Sep 2;184(18):4713-4733.e22. doi: 10.1016/j.cell.2021.07.023. Epub 2021 Jul 23. PMID: 34352228; PMCID: PMC8299217.
[11]Mathew D, Giles JR, Baxter AE, et al. Deep immune profiling of COVID-19 patients reveals distinct immunotypes with therapeutic implications. Science. 2020 Sep 4;369(6508):eabc8511. doi: 10.1126/science.abc8511. Epub 2020 Jul 15. PMID: 32669297; PMCID: PMC7402624.
[12]Sadarangani, M., Marchant, A. & Kollmann, T.R. Immunological mechanisms of vaccine-induced protection against COVID-19 in humans. Nat Rev Immunol 21, 475-484 (2021).
[13]Kuhlmann C, Mayer CK, Claassen M, et al. Breakthrough infections with SARS-CoV-2 omicron despite mRNA vaccine booster dose. Lancet. 2022 Feb 12;399(10325):625-626. doi: 10.1016/S0140-6736(22)00090-3. Epub 2022 Jan 18. Erratum in: Lancet. 2022 Feb 12;399(10325):628. PMID: 35063123; PMCID: PMC8765759.
[14]Kanji JN, Zelyas N, MacDonald C, et al. False negative rate of COVID-19 PCR testing: a discordant testing analysis. Virol J. 2021 Jan 9;18(1):13. doi: 10.1186/s12985-021-01489-0. PMID: 33422083; PMCID: PMC7794619.
[15]Shao W, Guo T, Toussaint NC, et al. Comparative analysis of mRNA and protein degradation in prostate tissues indicates high stability of proteins. Nat Commun. 2019 Jun 7;10(1):2524. doi: 10.1038/s41467-019-10513-5. PMID: 31175306; PMCID: PMC6555818.
[16]Xiao Q, Zhang F, Xu L, et al. High-throughput proteomics and AI for cancer biomarker discovery. Adv Drug Deliv Rev. 2021 Sep;176:113844. doi: 10.1016/j.addr.2021.113844. Epub 2021 Jun 26. PMID: 34182017.