本文转载自“BioArt”。
1942年,C.H. Waddington在研究细胞发展命运时, 首次提出了表观遗传学的概念【1】。由于技术及生物体本身特点等原因, 目前研究较多的表观遗传机制主要集中在DNA甲基化和组蛋白修饰。目前,5-甲基胞嘧啶(5-methylcytosine, 5mC)和5-羟甲基胞嘧啶(5-hydroxymethylcytosine, 5hmC)是哺乳动物基因组中发现的两种最常见的表观遗传标记。细胞通过双加氧酶家族TET (ten-eleven translocation)蛋白将5mC氧化为5hmC,随后进一步将5hmC氧化成5-甲酰基胞嘧啶(5-formylcytosine, 5fC)和5-羧基胞嘧啶(5-carboxylcytosine, 5caC)。与5mC和5hmC相比,5fC和5caC在哺乳动物基因组中的含量极低。DNA中胞嘧啶修饰从基因调控到生长发育过程都起着至关重要的作用【2】,异常的DNA甲基化和羟甲基化与各种疾病有关,因此确定5mC和5hmC的基因组分布对于临床应用意义重大【3-4】。

目前,DNA甲基化和羟甲基化分析测序行业的黄金标准是亚硫酸氢盐测序【5-6】及其衍生方法:TET辅助亚硫酸氢盐测序(TET-assisted bisulfite sequencing, TAB-Seq)【7】和氧化亚硫酸氢盐测序(oxidative bisulfite sequencing, oxBS) 等【8】。这些方法都采用亚硫酸氢盐处理将未甲基化的胞嘧啶转化为尿嘧啶,同时保持5mC和/或5hmC完整,随后的PCR扩增将尿嘧啶转化为胸腺嘧啶,因而实现在单碱基水平推断每个胞嘧啶的修饰。然而,亚硫酸氢盐测序存在两个主要缺点:1)亚硫酸氢盐处理条件过于剧烈,DNA可能会降解从而降低PCR 及后续分析技术的灵敏度;2)亚硫酸氢盐测序将未修饰的胞嘧啶完全转化为胸腺嘧啶(未修饰的胞嘧啶占人类基因组中总胞嘧啶的约95%),将所有胞嘧啶转化为胸腺嘧啶严重降低了序列复杂性,导致测序质量差,定位率低,基因组覆盖不均匀和测序成本增加。
为了解决以上问题,近日牛津大学宋春晓课题组等在Nature biotechnology发表题为Bisulfite-free direct detection of 5-methylcytosine and 5-hydroxymethylcytosine at base resolution的文章,开发了一种无需亚硫酸氢盐的测序方法:TET辅助吡啶硼烷测序(TET-assisted pyridine borane sequencing, TAPS)。利用TET将5mC和5hmC氧化为5caC,随后吡啶硼烷将5caC还原为二氢尿嘧啶(dihydrouracil, DHU),而后的PCR再将DHU转化为胸腺嘧啶。实例应用结果表明,与亚硫酸氢盐测序相比,TAPS可以实现更高的绘图速率,更均匀的覆盖率和更低的测序成本,从而实现更高的质量,更全面和更便宜的甲基化组分析。
在这项研究中,研究人员将含有11mer 5caC的DNA寡核苷酸作为模型DNA,筛选到了商业上可获得的环境友好型还原剂吡啶硼烷,确认其将5caC转化为DHU,同时吡啶硼烷也可通过脱甲酰化/脱氨基作用将5fC转化为DHU,对未甲基化胞嘧啶、5mC或5hmC都没有活性。TAPS过程利用TET将5mC和5hmC氧化为5caC,随后吡啶硼烷将5caC还原为DHU,而后的PCR再将DHU转化为胸腺嘧啶,因此可以用于5mC和5hmC的碱基水平检测。
此外,研究人员还进一步开发TAPSβ的过程:利用β-葡萄糖基转移酶标记5hmC使其糖基化,从而仅选择性测序5mC,然后从TAPS结果中减去TAPSβ来推断5hmC位点。研究人员还使用过氧化钾取代TET,将5hmC特异性氧化成5fC,可用于特异性地测序5hmC,研究人员将此方法被命名为:化学辅助吡啶硼烷测序(chemical-assisted pyridine borane sequencing, CAPS)。因此,TAPS和相关方法原则上可以对所有表观遗传修饰的胞嘧啶进行测序(如下图)。

图1硼烷还原DNA寡核苷酸
为了检测TAPS的性能,本文将其应用于小鼠胚胎干细胞(mESCs)基因组DNA的测序。HPLC-MS/MS定量显示,修饰型胞嘧啶中5mC占98.5%,其余部分由5hmC(1.5%)和痕量的5fC和5caC组成,没有DHU。在TET氧化后,约96%的修饰型胞嘧啶被氧化为5caC,3%被氧化为5fC。硼烷还原后,超过99%的修饰型胞嘧啶转化为DHU。这些结果表明,TET氧化和硼烷还原均有效地对基因组DNA起作用。TET氧化和硼烷还原均为温和反应,与亚硫酸氢盐处理相比没有显著的DNA降解,且DNA在TAPS后仍然是双链的,并且转化与DNA长度无关。

图2模板DNA和mESC基因组DNA的TAPS
随后研究人员对来自E14 mESCs23的两个基因组DNA样品进行了全基因组测序,一个使用TAPS(100 ng DNA),另一个使用标准全基因组亚硫酸氢盐测序(WGBS)(200 ng DNA)。结果,mTet1和吡啶硼烷的组合中5mC达到最高的转化率而未修饰胞嘧啶的转化率最低(图3a, b),5hmC的转化率比5mC低8.2%,非CpG环境的转化率比CpG环境低11.4%(图3a),与亚硫酸氢盐测序相当。表明TAPS在5mC和5hmC存在时都能很好地工作。WGBS数据处理需要特殊软件(如Bismark25),而TAPS使用名为“asTair”的自定义修改调用工具,当处理模拟的WGBS和TAPS读数时,TAPS / asTair比WGBS / Bismark快三倍以上(图3c)。由于WGBS文库中几乎所有胞嘧啶转化为胸腺嘧啶,极其偏差的核苷酸构成会对Illumina测序产生负面影响,导致WGBS读数显示胞嘧啶/鸟嘌呤碱基对的测序质量得分比TAPS显著降低(图3d)。而亚硫酸氢盐处理后DNA会降解,使得WGBS的定位率比TAPS显著降低(图3e)。因此,对于相同的测序成本,TAPS的平均测序深度超过WGBS的平均深度。

图3 TAPS测序质量优于WGBS
在WGBS相同的测序深度下,TAPS未覆盖区域较少,并总体上显示更均匀的覆盖分布。即使控制WGBS和TAPS之间测序深度的差异,CpG岛(CGIs)也可被TAPS更好的覆盖(图4a)。在E14基因组所有42,741,339个CpG位点中,使用TAPS时88.3%被覆盖,而WGBS仅为77.6%(图4b)。TAPS和WGBS对跨染色体甲基化测序有良好的一致性(图4c)。在每个核苷酸的碱基上,两种方法都至少读数三次并有32,428,076个CpG位点重叠(图4b)。研究人员将。在WGBS读取的修饰的CpG位点中(修饰的CpG为修饰水平>10%的CpG),96.2%被TAPS修饰后读取,表明WGBS和TAPS之间具有良好的一致性(图4d)。而每个CpG的修饰水平中,WGBS和TAPS也存在良好的相关性(图4E)。这些结果表明TAPS可直接取代WGBS,且提供了比WGBS更全面的甲基化数据。最后,研究人员证明TAPS只需1 ng的基因组DNA即可完成实验。
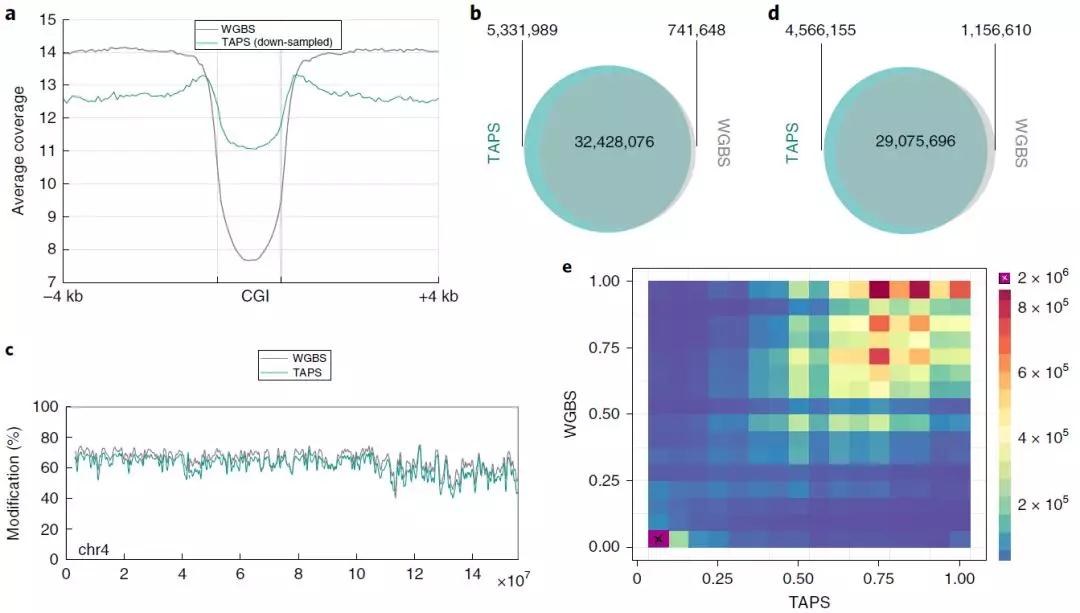
图4 TAPS和WGBS的全基因组甲基化比较
综上,研究人员开发并证明TAPS可用于基因组甲基化测序,可在常规使用中直接替代WGBS,同时降低分析成本并提供高质量和更完整的甲基化组。TAPS与各种下游分析方法兼容(如:焦磷酸测序、限制性消化、MALDI质谱、微阵列和全基因组测序等),并能保留长链DNA,因此与SMRT测序和纳米孔测序等长链测序技术结合,可用于研究某些难以测序的区域,提高其应用价值。
值得一提的是,数月前,来自北京大学生科院的伊成器实验室就在 Journal of the American Chemical Society杂志上发表了题为Bisulfite-Free, Nanoscale Analysis of 5-Hydroxymethylcytosine at Single Base Resolution的论文(https://pubs.acs.org.ccindex.cn/doi/10.1021/jacs.8b08297),首次实现了免亚硫酸氢盐(bisulfite free)的5-羟甲基胞嘧啶(5hmC)全基因组单碱基分辨率测定,并且解析了之前开发的系列核酸修饰测序技术的分子机理。

该研究基于5hmC的特异性氧化与化学标记,且标记产物在随后的PCR扩增过程发生C到T转变的现象,开发了免亚硫酸氢盐的5hmC全基因组单碱基分辨率测序技术(hmC-CATCH)。除了hmC-CATCH技术外,伊成器课题组与合作者还开发了系列免亚硫酸氢盐的核酸修饰测序技术(Xia et al., Nature Methods 2015; Zhu et al., Cell Stem Cell 2017),这些测序技术都是基于化合物特异性标记5-醛基胞嘧啶(5fC),生成非天然胞嘧啶碱基(M-fC或I-fC),并且这两个碱基在随后的PCR扩增过程实现C到T转变。
针对最新发表在NBT上的这一工作,BioArt采访了伊成器研究员,他表示:“现有的5-甲基胞嘧啶测序技术依赖于亚硫酸氢盐处理,带来严重的DNA降解。这一新技术的开发,为5mC的检测提供了重要工具。该技术将5-甲基胞嘧啶的氧化与5-醛基胞嘧啶的特异性标记结合,极具创新性。这项技术与亚硫酸氢盐测序方法相比,在测序起始量、测序成本和测序数据质量等方面都显示出优越性,有望替代亚硫酸氢盐测序成为DNA甲基化检测新的“金标准”。”
参考文献:
1. Waddington CH. The epigenotype. 1942. Int J Epidemiol, 2012, 41(1): 10-13.
2. Li, E. & Zhang, Y. DNA methylation in mammals. Cold Spring Harb. Perspect. Biol. 6, a019133 (2014).
3. Chan, K. C. et al. Noninvasive detection of cancer-associated genome-wide hypomethylation and copy number aberrations by plasma DNA bisulfite sequencing. Proc. Natl Acad. Sci. USA 110, 18761–18768 (2013).
4. Song, C. X. et al. 5-Hydroxymethylcytosine signatures in cell-free DNA provide information about tumor types and stages. Cell Res. 27, 1231–1242 (2017).
5. Lister, R. et al. Global epigenomic reconfiguration during mammalian brain development. Science 341, 1237905 (2013).
6. Raiber, E.-A., Hardisty, R., van Delft, P. & Balasubramanian, S. Mapping and elucidating the function of modified bases in DNA. Nat. Rev. Chem. 1, 0069 (2017).
7. Yu, M. et al. Base-resolution analysis of 5-hydroxymethylcytosine in the mammalian genome. Cell 149, 1368–1380 (2012).
8. Booth, M. J. et al. Quantitative sequencing of 5-methylcytosine and 5-hydroxymethylcytosine at single-base resolution. Science 336, 934–937 (2012).