1.home网址:https://www.kegg.jp/kegg/
2.关于kegg数据库的一些统计情况:https://www.kegg.jp/kegg/docs/statistics.html截止2018.4.12该数据库总共包含:525个代谢通路(Pathway maps),21,952条同源群(KEGG Orthology (KO) groups),涉及物种442真核生物、4654细菌,268古细菌和317种病毒。涉及到基因数目26,161,327条。
3.区分map00010、ko00010、hsa00010
map:参考参考代谢通路图(图1),
ko:高亮过KOs的参考代谢通路图(图2紫色表示)
单属于人的代谢通路图以绿色标注出(图3绿色)
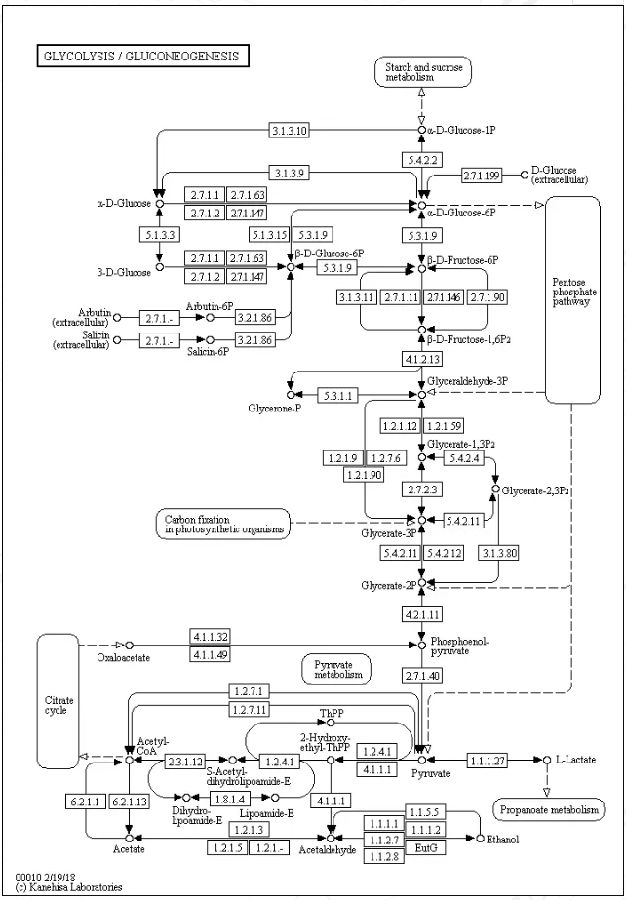
图 1代谢通路图map00010
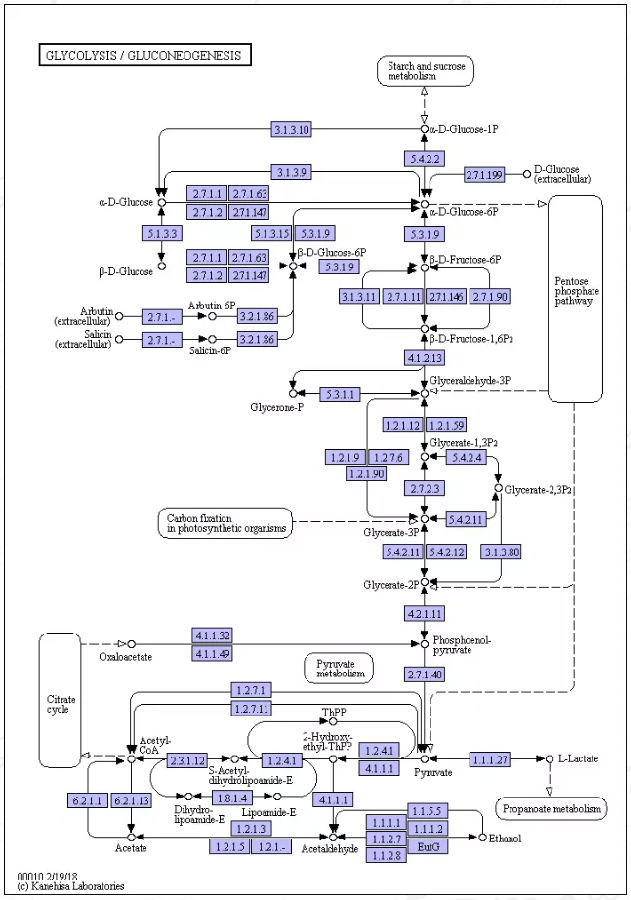
图 2高亮KO后的map00010

图 3 人的map00010
4. 特殊的代谢通路以及备注
(1)011与012:编码以011或012开头的代谢通路图为一些整合性质的代谢通路图,总共包含9个。
(2)010:是化学结构图并没有新的KO扩展
(3)07:与药物结构相关的代谢通路图,并没有新的KO扩展
(4)常规基因kegg数据库注释分析,就是分析ko中去除011、012、010以及07开头的代谢通路,总共431条目。
(5)ko与KO的区别:ko号码是KEGG中一类参考代谢通路,而KO代表的是一类具有相似功能的基因簇。
5.在kegg数据库收费的情况如何实现对基因序列的批量注释?
(1)通过KEGG的API,首先你可以获得KEGG数据库中所有物种简写列表:https://rest.kegg.jp/list/organism
(2)使用kobas软件,与koabs数据库做比对注释,kobas可以选择对应的参考物种,如果是未知物种可以选择ko,从kobas的输出结果中你可以获得你所注释的基因与kegg数据库中的geneID的对应关系
(3) 以人(hsa)为例,通过KEGG的API你可以获得你关心物种的基因geneID所对应的代谢通路也就是ko:https://rest.kegg.jp/link/pathway/hsa
(4)以人(hsa)为例,通过KEGG的API你可以获得你关心物种的基因geneID所在的同源群也就是KO号: https://rest.kegg.jp/link/ko/hsa
(5)最后结合kobas得到的query与geneID以及kegg数据库中得到的geneID与KO, geneID与ko之间的关系,就可以完整的到一组未知基因的kegg数据库注释了。
-END-
本文系欧易生物原创
欢迎转发到朋友圈
转载请注明本文转自欧易生物