2018年3月,赵立平教授和团队在《科学》杂志上发表了一篇论文,利用多种膳食纤维的组合,改善了糖尿病人的肠道菌群,选择性地促进一组产短链脂肪酸细菌的富集。研究发现这组菌的丰度和多样性越高,血糖改善越好。[1]
这篇文章中利用宏基因组测序,在常规宏基因组分析的基础上,进一步使用了contig binning方法,充分利用了宏基因组数据,挖掘出有用信息。
contig binning究竟是一种什么样的神器呢?今天就和各位详细道来。
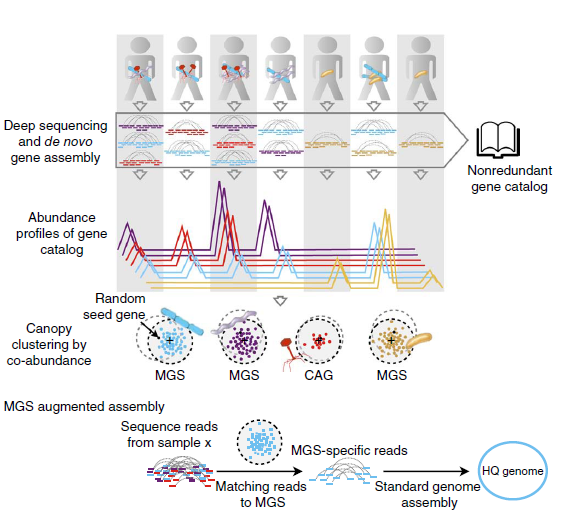
我们先来看看欧易生物宏基因组-contig binning的分析流程吧:

如上图所展示,contig binning在常规宏基因组基础上,进一步将拼接的contig进行cluster聚类,可实现单菌的组装以及宏基因组关联分析(MWAS)分析。
- 1.contig binning来源和进展 -
虽然微生物的培养技术已经发展了很多年,但是环境中可培养的微生物比例仍然较低。而对于微生物的研究,往往是需要精确到菌株水平的。
基于宏基因组数据的contig binning分析,可基于宏基因组组装结果,将组成相似以及丰度分布模式一致的contig划分到同一物种,并进一步进行单菌的草图组装。从而可在基于单菌组装结果的基础上进行菌株水平的基因和功能注释、比较基因组分析、进化分析等。
- 早在2011年,发表于Science上的一篇基于牛瘤胃宏基因组的研究最早用到了binning分析。该研究从268G的宏基因数据中成功binning出了15个不能培养的微生物的全基因组序列。并且最终使用单细胞全基因组测序的方法进行了验证。[2]
- 2013年发表于Nature Biotechnology的一篇文章中,作者利用污泥的29+59G数据量重建了31个基因组,包含一些低丰度的物种,也包含了不能分离培养的TM7门类。[3]
- 2014年发表于Nature Communication的一篇文章中,作者利用20G的宏基因组数据,在季节性融化冻土层土壤中组装了1个高质量的产甲烷菌基因组。该文章使用的contig binning分析软件为bio-kmer counter。[4]
- 2015年发表于Nature的一篇研究北极圈冻土层和活跃土壤的文章,作者利用84.2G的宏基因组数据以及20.4G的宏转录组数据,最终得到22个高质量的bins。[5]
- 2016年发表于Biotechnol Biofuel的一篇文章,从沼气池发酵液、牛瘤胃和大象的粪便中测序得到了131G的数据,最终完成了104个高质量基因组的bins。文章中使用的contig binning分析软件为CONCOCT。[6]
- 2017年瑞典乌普萨拉大学的一个国际研究小组在Nature上发表的一项新研究中,通过binning方法,对于无法在实验室培养的微生物基因组数据,确定了一组新的古细菌。[7]
- 2018年3月份,赵立平教授团队发表在Science上的一篇文章,利用了contig binning分析,定位到15个菌株,这15个菌株是高膳食纤维改善代谢的主要贡献者。[1]
- 2.contig binning原理 -
最开始进行binning分析的依据是,来自同一菌株的序列,其核酸组成是相似的。于是可以根据核酸组成信息来进行binning,例如根据核酸使用频率(oligonucleotide frequency variations),通常是四核苷酸频率(tetranucleotide frequency)、GC含量和必需的单拷贝基因等。即根据核酸组成(NC-Nucleotide composition)来进行contig binning。
随后的研究发现来自同一个菌株的基因在不同的样品中 ( 不同时间或不同病理程度 ) 的丰度分布模式是相似的 (Nielsen et al., 2014)。[8]
因此可以根据基因在不同样品中的丰度变化模式( co-abundance patterns across multiple samples),即微分丰度(DA-Differential abundance)来进行contig binning。这种方法更有普适性,一般效果也比较好,能达到菌株的水平。
但这种方式需要较大样本量,一般至少要50个样本以上,且至少要有2个组能呈现丰度变化 ( 即不同的处理、不同的时间、疾病和健康、或者不同的采样地点等 ) ,每个组内的生物学重复也要尽量的多。
还可以同时依据核酸组成和丰度变化信息,即核酸组成与丰度(NCA-Nucleotide composition and abundance),利用核酸组成信息和丰度差异综合计算距离矩阵,既能保证binning效果,也能相对节约计算资源,现在比较主流的binning软件大多是NCA算法。
- 3.contig binning软件的评估 -
Microbiome期刊于2016年发表了一篇综述,认为contig binning的组装策略主要分为3类:核酸组成(NC)、核酸组成与丰度(NCA)、微分丰度(DA)等,然而各个方法都有各自的优缺点(Sangwan et al., 2016)[9]。不同的contig binning涉及到的软件以及具体信息如下表所示:
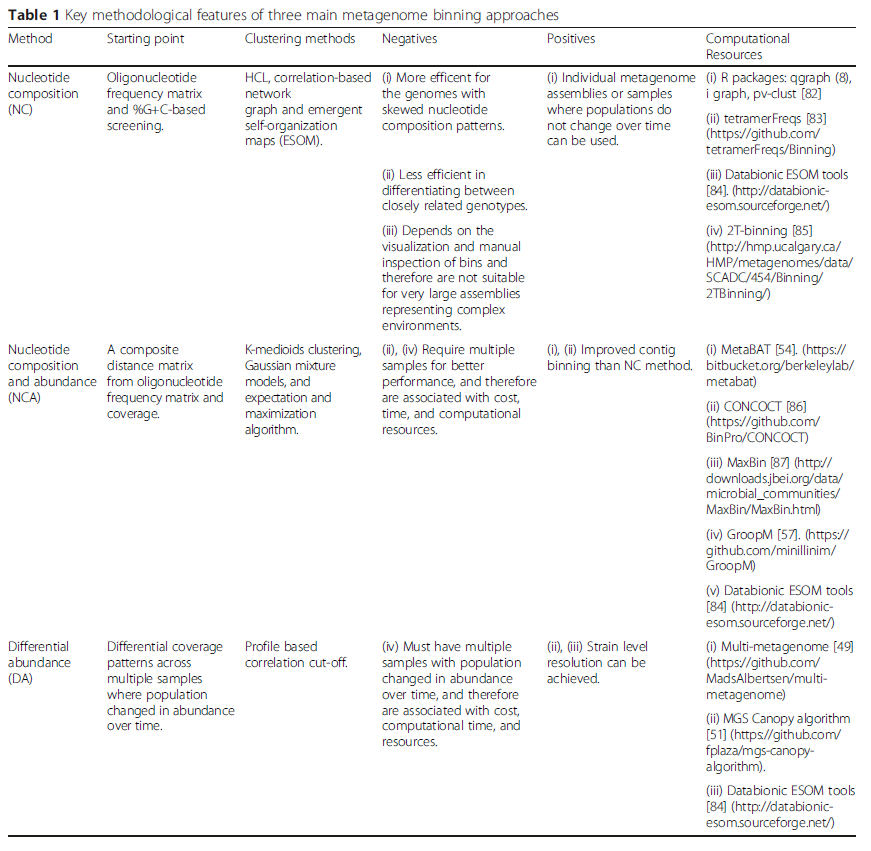
对于常见的一些 NCA-based contig binning 软件,文章也做了以下比较:
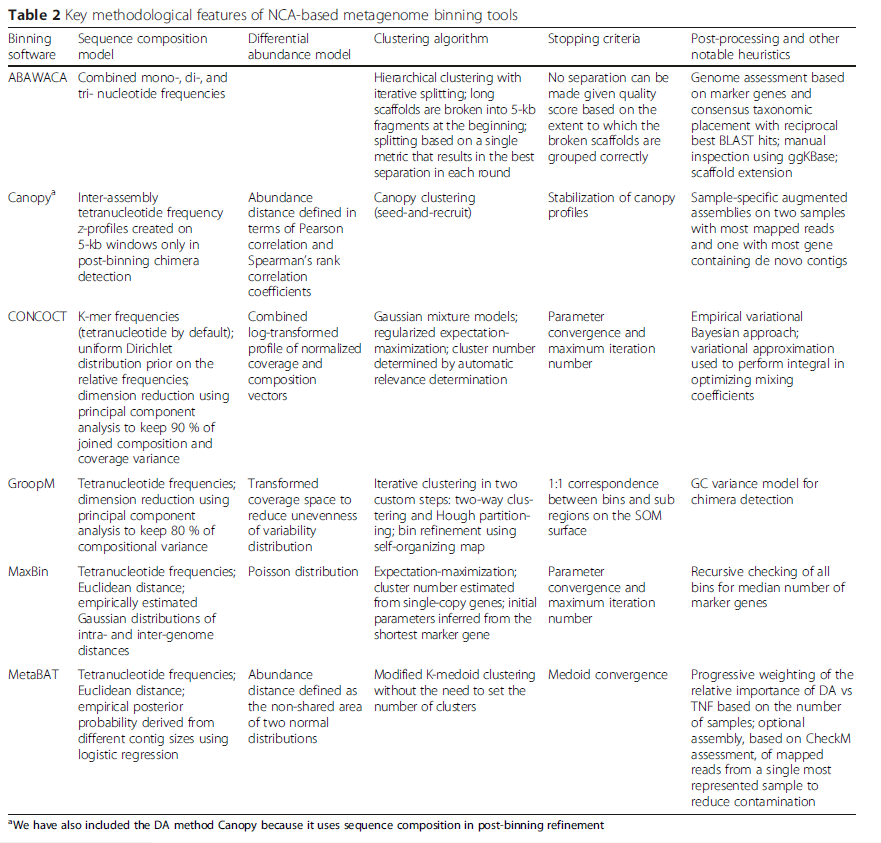
- 4.contig binning分析方法的应用 -
宏基因组中引用contig binning分析方法,主要有两方面应用:宏基因组关联分析(MWAS-Metagenome Wide-Association Study)以及单菌组装。
4.1 宏基因组关联分析(MWAS)
通过binning得到的bins(strain-level clusters 或strain-level taxonomic units)可以进行宏基因组关联分析(MWAS/MGWAS: Metagenome-wide association studies) 以及多组学联合分析,将特定功能代谢产物与特定物种、特定基因进行关联研究。探究疾病的因果机制,为疾病监控、环境监测提供了菌株水平的生物靶标。
根据具体的聚类算法和相关性系数的不同,对binning得到的bins的命名也不同。主要有MLG ( metagenomic linkage groups), MGC ( metagenomic clusters )以及CAG ( co-abundance gene groups )。其中将包含有700个以上的gene的CAG称为MGS(metagenomic species)。以及MetaOTUs ( metagenomic operational taxonomic units )[10]。MLG, MGC, MGS和MetaOTUs在物种注释的标准根据具体的文章算法可能不同。
2016年在Nature Reviews Microbiology发表的一篇MWAS的综述中(Wang, 2016) [11],详细介绍了已发表的一些疾病在宏基因组binning研究中用到的binning方法。
Qin在2013年发表于Nature的一篇有关II型糖尿病的宏基因组研究,就是使用了MLG(metagenomic linkage groups)分析方法,进行了与疾病相关的MWAS分析。并使用了PERMANOVA方法计算相关性。[12]
而对于宏基因组和疾病的关联分析,常见的一些关联分析方法如下:
多元统计分析:例如根据PERMANOVA分析,识别和分组显著相关的因素(例如药物干预或者疾病);
非监督模型聚类分析: 如肠型分析;
差异检验:根据差异检验统计算法,识别组间差异物种或差异基因;
功能分析: 识别和疾病等相关的通路或者功能;
分类模型构建: 例如使用决策树算法或者分类算法,对分类模型构建并评估。
4.2 基因bins的单菌组装
一般情况下,宏基因组测序由于其组装难度,以及存在大量的未知物种,最终可有效利用的数据量比例较低。而contig binning却能够很好的利用到这些数据,最大限度地得到菌群的组成信息,虽然得到的菌群可能是未知物种。
Fredirk的研究发现,用MGS数据构建的模型,和仅用已知菌群构建的模型相比,对糖尿病诊断的AUC面积可以从0.71提高到0.83 。
针对bins的单菌组装的方法和步骤如下:
Step1: 得到bins后的组装
将各个样品的clean reads分别同该bin所包含的contigs进行比对,并计算每个样品map上的比例,选取map率高的前几个样品map上的reads分别单独组装,同时也可以将map率高的这几个样品中map上的reads混合在一起进行组装,可选取组装效果好的做为最终组装结果。
对于组装软件,可使用常用的组装软件SOAPdenovo、Velvet、ABySS、Spades 等。
Step2: 组装效果评估
对于上述bins重新组装得到的基因组草图,可以进行基因组完整度的评估,也可以用CheckM和GC-Depth分布图,来评估组装效果。
Step3: 组装的单菌基因组分析
对组装后的基因组草图,可进一步进行单菌组装出的基因组分析,包括基因组组分分析、基因预测以及功能注释分析、共线性比较分析等。
如果需要对组装出来的单菌进行注释,可以基于NR库。后续可进行ANI( average nucleotide identity )等分析。种内菌株的精细化鉴定还可以借助系统发育分析,比较基因组分析等方法。
由于组装出来的基因组草图很可能是未知的物种,在NCBI中并没有近缘相关的参考基因组,或者bin的基因组草图组装的太碎,都可能导致物种鉴定达不到理想的效果。
关于contig binning的介绍基本差不多了,看到这里有没有很心动?
如果各位老师手头有大量的宏基因组数据,却不知道如何有效利用,或希望通过宏基因组测序分析进一步精确到菌株水平的研究,现在就可以快快联系我们了!
- 参考文献 -
[1] Zhao L P, Zhang F, Ding X Y, et al. Gut bacteria selectively promoted by dietary fibers alleviate type 2 diabetes.[J]. Science, 2018, 1151-1156.
[2] Hess, M. et al. Metagenomic discovery of biomass-degrading genes and genomes from cow rumen. Science, 2011, 463-467.
[3] Albertsen M, Hugenholtz P, Skarshewski A, et al. Genome sequences of rare, uncultured bacteria obtained by differential coverage binning of multiple metagenomes[J]. Nature biotechnology, 2013, 31(6): 533-538.
[4] Mondav R, Woodcroft B J, Kim E H, et al. Discovery of a novel methanogen prevalent in thawing permafrost[J]. Nature Communications, 2014, 5(2):3212.
[5] Hultman J, Waldrop M P, Mackelprang R, et al. Multi-omics of permafrost, active layer and thermokarst bog soil microbiomes.[J]. Nature, 2015, 521(7551):208-12.
[6] Güllert S, Fischer M A, Turaev D, et al. Deep metagenome and metatranscriptome analyses of microbial communities affiliated with an industrial biogas fermenter, a cow rumen, and elephant feces reveal major differences in carbohydrate hydrolysis strategies[J]. Biotechnology for Biofuels, 2016, 9(1):121.
[7] Zarembaniedzwiedzka K, Caceres E F, Saw J H, et al. Asgard archaea illuminate the origin of eukaryotic cellular complexity.[J]. Nature, 2017, 541(7637):353.
[8] Nielsen H B, Almeida M, Juncker A S, et al. Identification and assembly of genomes and genetic elements in complex metagenomic samples without using reference genomes.[J]. Nature Biotechnology, 2014, 32(8):822-828.
[9] Sangwan N, Xia F, Gilbert J A. Recovering complete and draft population genomes from metagenome datasets[J]. Microbiome, 2016, 4(1):8.
[10] Bäckhed F, Roswall J, Peng Y, et al. Dynamics and Stabilization of the Human Gut Microbiome during the First Year of Life.[J]. Cell Host & Microbe, 2015, 17(5):690-703.
[11] Wang J, Jia H. Metagenome-wide association studies: fine-mining the microbiome[J]. Nature Reviews Microbiology, 2016, 14(8):508-522.
[12] Qin J, Li Y, Cai Z, et al. A metagenome-wide association study of gut microbiota in type 2 diabetes[J]. Nature, 2013, 490(7418):55.
- END -
本文系欧易生物原创
欢迎转发到朋友圈
转载请注明本文转自欧易生物