BLAST是NCBI最重要的生物信息学工具,但BLAST里面涉及的知识比较多,今天就来简单的介绍一下这款功能强大的工具,你准备好了吗?
什么是BLAST
BLAST(Basic Local Alignment Search Tool,局部相似性基本查询工具)是由美国生物技术信息中心NCBI开发的一个基于序列相似性的数据库搜索程序,是目前序列比对中功能和数据库最为全面的,因而数据结果更容易得到期刊编审的认可,在生信分析中有着广泛的应用。
功能:蛋白或核酸序列与生物数据库的比较;
序列(查询序列)与序列中所有的生物序列进行成对序列比对;
为什么使用BLAST?
BLAST是快速、准确并可通过网络访问的生物信息学工具。(利用了“启发式”(heuristic)算法)
可以快速地完成对大容量的生物序列数据库的捜索。
BLAST的用途
确定查询序列是否有已知的直系同源或旁系同源类似物;
确定一个特定基因或蛋白的突变体;
研究EST (Expressed sequence tags)是否存在选择性剪接;
寻找对蛋白质结构和(或功能)重要的残基或结构域
……
初步了解了BLAST之后,我们来进一步熟悉BLAST
线上BLAST:
Blast的线上主页为:https://blast.ncbi.nlm.nih.gov/Blast.cgi
经过多次升级改版之后,blast提供了多种比对方式和丰富的“Specialized searches”。
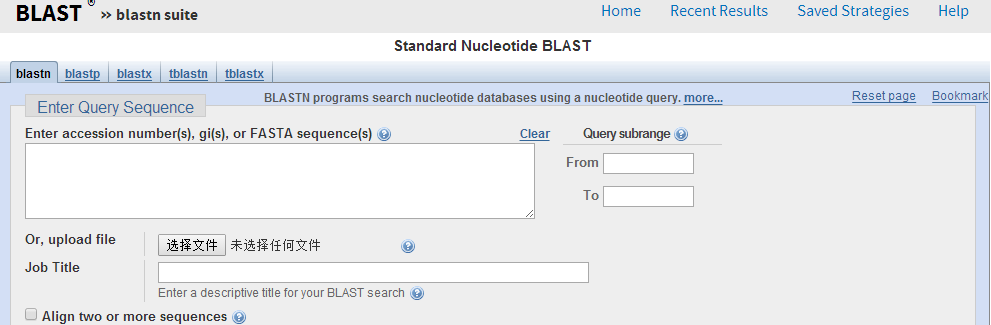
Basic BLAST 提供了5种Blast:
Blastn:将待查询的核酸序列及其互补序列一起对核酸序列数据库进行查询。
Blastp:将待查询的蛋白质序列及其互补序列一起对蛋白质序列数据库进行查询。
Blastx:是一种以核酸序列查询蛋白质序列的方式。先将带查询的核酸序列按六种可读框架(逐个向前三个碱基和逐个向后三个碱基读码)翻译成蛋白质序列,然后将翻译结果对蛋白质序列数据库进行查询。
tblastn:以蛋白质序列查询核酸库。先将核酸序列数据库中的核酸序列按六种可读框架翻译成蛋白质序列,然后将待查询的蛋白质序列及其互补序列对其翻译结果进行查询。
tblastx:以核酸查询核酸库。先将待查询的核酸序列和核酸序列数据库中的核酸序列按六种可读框架翻译成蛋白质序列,然后再将两种翻译结果从蛋白质水平进行查询。
线上Blast一般一次只能比对一条序列,当我们需要对大规模的数据做分析或者研究的序列本身具有保密性的话,我们可以将BLAST下载到本地,网站主页也提供了这样的功能。

点进Download BLAST就可以在目录中选择最新的BLAST版本进行下载,或者直接使用链接:ftp://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/
可以选择安装版或者解压版。

本地BLAST:
安装或者解压完成后会有两个子目录,“Bin”和“doc”,其中“bin”目录下,将BLAST的基础功能以EXE可执行文件的方式实现。
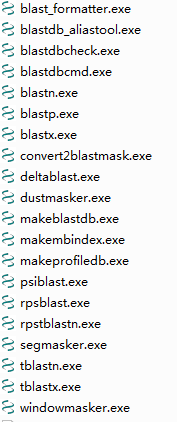
可以将“bin”目录的地址放入环境变量中,就可以在任意目录下使用BLAST中的功能。
数据库的获取:
可以从NCBI、Ensemble、UniPort等数据库网站下载序列信息作为库文件(fasta格式)。

下载后结果如下图,在后续操作中无需改动。

在命令行模式下使用“makeblastdb.exe”来创建数据库。
可以先输入命令行
Makeblastdb –help
来查看makeblastdb的功能及对相关参数的要求。
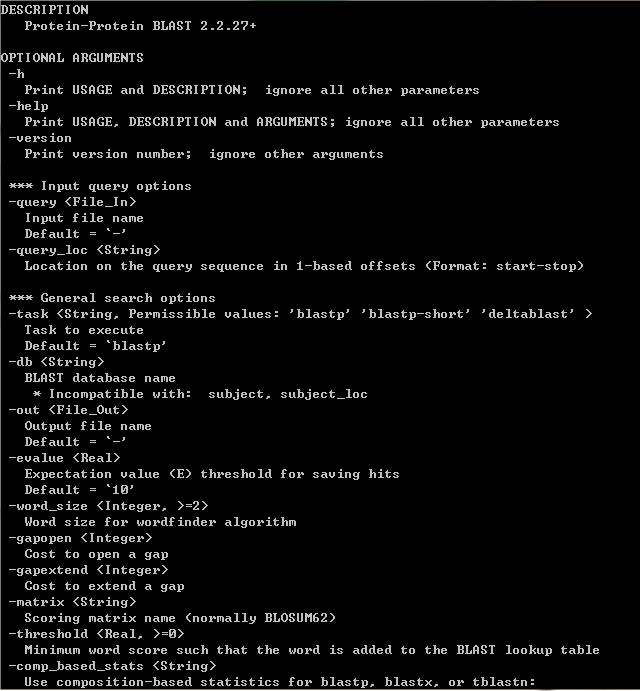
在使用中我们不必考虑所有的参数,只要能够准确输入作为数据库的fasta文件以及部分参数,就能够获取所需的数据库。
-dbtype:输入字符串,“nucl”表示核酸序列,“port”表示蛋白序列。
-in :输入要用作数据库的fasta 文件。
-out:要创建的数据库的名字
这里我们以用作演示的序列eg.fasta来创建库。
命令行示例:
makeblastdb -in eg.fasta -dbtype prot -out database
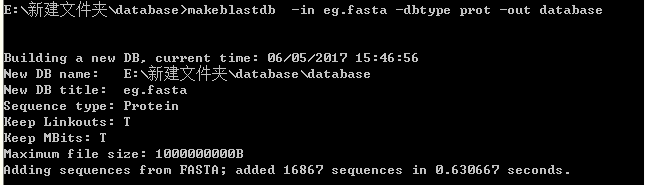
对于比较小的库,不到一秒钟就能完成建库。运行结果会产生database.pin,database.phr,database.psq三个格式的文件。
比 对:
本地比对中,我们可以将所有要分析的序列一特定的格式存入fasta文件中,然后对整个文件进行分析。
![]()
blastx-query query.fasta -db database -out results.txt -evalue 1e-5 -outfmt 6-perc_identity 90 -max_target_seqs 5 -num_threads 2
其中参数:
-query:输入要分析的文件路径及文件名,默认路径下可以只输入文件名
-out:输出文件路径及文件名
-db:数据库路径及数据库名
-evalue:设置输出结果的e-value值,一般设为1e-5
-max_target_seqs:最多允许比对到数据库中的序列数目,参数仅适用于outfmt >4。 -perc_identity :比对的最低相似度
-num_threads:线程数,线程越多,运行速度越快,但占用的CPU也越多,不能高于电脑的线程数。
-outfmt:输出文件格式,总共有15种,一般设置为6。6是tabular格式对应BLAST的m8格式,其输出结果示例为:
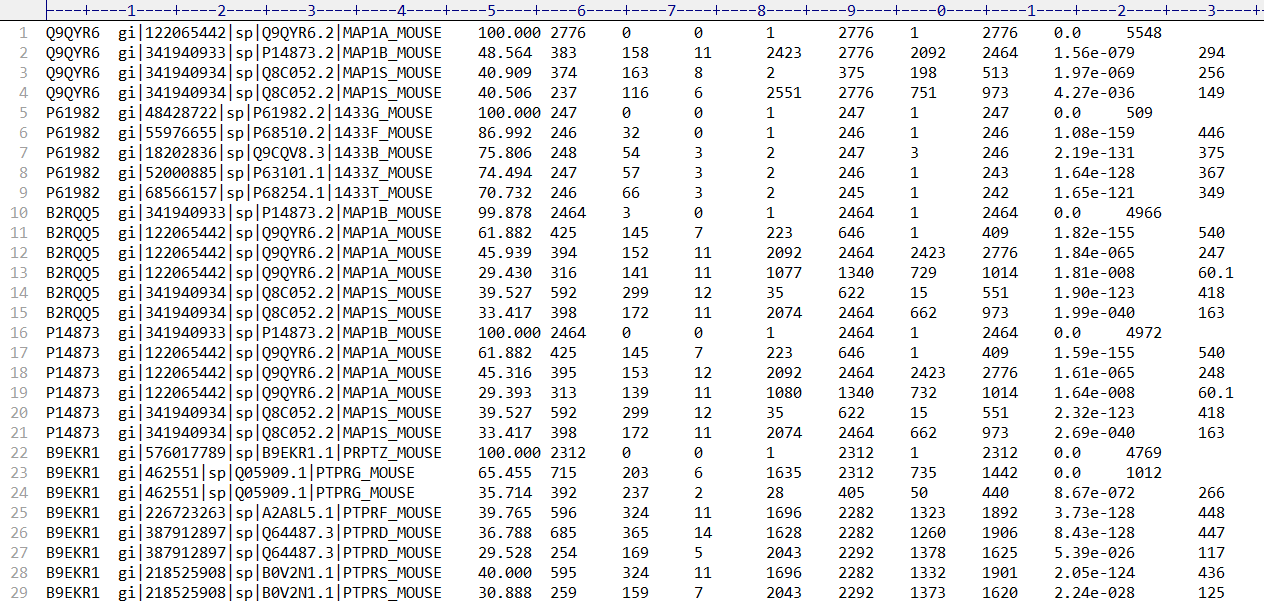
以下是各个数字所代表的输出格式。
0 =pairwise,
1 =query-anchored showing identities,
2 =query-anchored no identities,
3 =flat query-anchored, show identities,
4 =flat query-anchored, no identities,
5 =XML Blast output,
6 =tabular,
7 =tabular with comment lines,
8 =Text ASN.1,9 = Binary ASN.1,
10= Comma-separated values,
11= BLAST archive format (ASN.1),
12= JSON Seqalign output,
13= JSON Blast output,
14= XML2 Blast output
每种格式都有其适用的范围,需要根据之后的分析和用途来选择。
以上就是BLAST的入门教程,如果熟悉了BLAST的基本的操作,一些简单粗浅的对比就可以轻松应对啦~
什么!你还是搞不定?没关系,来找中科,中科新生命提供优质的生物信息学服务,我们帮你搞定!