十月初,《The Scientist》杂志为纪念创刊30周年,推出30年生命科学领域主流技术发展的回顾特刊。专题针对“DNA测序”、“显微镜”、“神经科学”、“基因编辑”和“干细胞”5大领域相关研究突破和技术变革展开论述,并以时间轴的形式呈现出科学发展的精彩和魅力。
生物探索小编对其中内容进行了编译,整理如下:
DNA测序

基因测序技术直接推动着生命医学的发展,并改变着我们对物种多样性、疾病机理和防治策略等多领域的认知和理解。过去,DNA测序技术成本高昂、繁琐费时。如今,它已经进入了快速低廉、且拥有标准体系的高通量时代,先后经历了3次变革(Sanger链终止法、高通量测序、单分子测序)。
上世纪70年代,DNA测序技术的研究开始绽放火花。初期,华裔分子生物学家吴瑞先生开创性设计出“引物延伸”的测序策略。1977年,剑桥大学生物化学家Frederick Sanger及其团队在引物延伸策略的基础之上,在PNAS发表文章,利用DNA聚合酶的双脱氧链终止原理创建了DNA测序主流技术——Sanger法(双脱氧链终止法)。
同年,美国哈佛大学物理学家与生物化学家Walter Gilbert与 Alan Maxam合作共同基于Sanger直读法原理,利用特异的化学试剂修饰不同碱基,独立提出另一种测序方法——化学裂解法。
传统的化学降解法、Sanger法以及在它们基础之上发展来的各种测序技术统称为第一代DNA测序技术。1986年美国应用生物系统公司ABI(Applied Biosystems Inc.成立于1981年)基于第一代测序技术推出世界首个自动化DNA荧光测序仪。1990年人类基因组计划(HGP)启动,并耗时10年完成人类基因组草图的绘制。
人类基因组计划的完成让我们进入功能基因组时代,无疑提高了对DNA测序技术的深度、速度和重复性等方面的高需求,从而推动了第二代测序技术的发展。2005年,454生命科学公司利用焦磷酸测序原理推出首个第二代测序平台,能够以99%的准确率完成2500万碱基序列的读取工作。
随后,以罗氏的GS FLX测序平台、Illumina的Solexa Genome Analyzer测序平台和ABI的SOLiD测序平台为代表的第二代测序技术先后在市场上崭露头角,它们的显著特点包括大规模平行测序、低成本,并因此得到广泛应用,例如分子诊断行业。
现在,以单分子测序为特征的第三代测序技术已经出现,典型的包括Helicos公司的Heliscope单分子测序仪、Pacific Biosciences公司的SMRT技术、Oxford Nanopore Technologies公司正在研究的纳米孔单分子技术。这一最新技术旨在克服错误率问题,使得测序不再需要PCR扩增环节,延续了高通量测序的优点并实现了更为快速、低廉的单分子测序。
基因测序是实现个性化医疗和精准医学的关键技术。未来,它还将面临着更大的发展和应用空间。
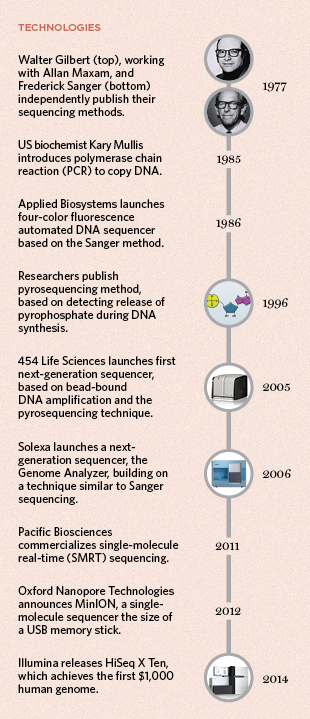
1977: Sanger—NIH, Gilbert—National Library of Medicine; 1996: TS Staff; 2005: Courtesy of Roche; 2006: Biochemical Society Transactions Feb 01, 2015,43(1)1-5; DOI: 10.1042/BST20140254; 2014: Courtesy of Illumina
干细胞

关于干细胞的研究可追溯至上世纪90年代。
1981年,科学家从小鼠胚胎中分离出多能干细胞,这一类特殊的细胞能够自我更新、复制出更多的细胞用于研究,同时还可以作为构架转基因小鼠的原材料。多伦多儿童医院的发育生物学家Janet Rossant表示:“老鼠胚胎干细胞改变了整个哺乳动物遗传学的研究。”
17年后,科学家能够将小鼠胚胎干细胞(ESCs)诱导分化成多种细胞类型,例如造血细胞、肌肉细胞和神经细胞。同时,他们开始将目光转向人类细胞。1998年,威斯康星-麦迪逊大学的发育生物学家James Thomson带领团队成功从捐赠的人类体外受精胚胎中分离出干细胞,同年约翰霍普金斯大学的John Gearhart团队也在捐赠的胎儿组织中成功获取胚胎干细胞。
1996年,有着克隆之父之称的胚胎学家Ian Wilmut团队成功培育出第一只克隆羊多莉。多莉的出生运用的是无性生殖技术,通过将乳腺细胞的细胞核移入已经去核的卵细胞中,使其融合形成胚胎细胞,并最终成功克隆出来小绵羊。这意味着,高度分化的成熟细胞也具有全能性的潜力。随后的细胞融合试验表明,人类胚胎干细胞也存在重编程因子,使其具有分化成多种细胞类型的能力。
但是,以胚胎为原材料展开的干细胞研究涉及伦理问题。那么,是否可以对体细胞重编程使其重返多能性呢?2006年,山中伸弥(Shinya Yamanaka)实验室成功利用病毒载体将4个转录因子转入成年老鼠的皮肤细胞中,使其重编程并获得一簇类似于胚胎干细胞(ESCs)的新型细胞,由此打开了诱导性多能干细胞(induced pluripotent stem cells,iPSCs)的研究大门。
2007年,包括山中伸弥在内的三个研究团队再次重复该试验并改进了重编程方法。同时,山中伸弥和威斯康星大学麦迪逊分校James Thomson合作,成功将人类成体细胞诱导成多能性干细胞,并因此入选Science年度十大科学突破。截止2009年,围绕诱导性多能干细胞的学术文章多达300篇。
诱导性多能干细胞与胚胎干细胞的类似程度有多少?已有研究证实,诱导性多功能干细胞依然保留着原成熟细胞的表观遗传学记忆。但是,专家认为这种差异不会影响细胞治疗的效果。
全球多个实验室正在多能性细胞生成应用于临床治疗的多种细胞,例如胰腺β细胞(糖尿病)、多巴胺神经细胞(帕金森症)。2010年,美国Geron公司获FDA批准首次开启基于人类胚胎干细胞治疗脊椎损伤的临床试验。遗憾的是,2011年,因为高额成本问题该项目被终止。
2014年,日本理化学研究所(RIKEN)干细胞临床研究员Masayo Takahashi团队完成首例诱导性多能干细胞治疗黄斑变性的临床试验,并取得成功。在第二例临床试验开战之前,出于安全等顾虑这一临床项目被暂停。但是,2016年6月,RIKEN研究所发布公告将重新启动基于异体iPS细胞治疗黄斑变性的临床试验。
基于干细胞的自我更新复制、具有多向分化的独特功能,其在转化医学、再生医学、精准医学的应用越来越受到重视。

1981: iStock.com/tiripero; 1998: Julie Baker lab, Stanford University School of Medicine, via CIRM; 2001: White House photo by Eric Draper; 2007: Kathrin Plath lab, University of California, Los Angeles, via CIRM; 2009: iStock.com/halduns; 2010: BruceBlaus/Wikimedia Commons; 2014: CSIRO/Wikimedia Commons
基因编辑

谈及基因编辑的发展需要从基因敲除说起。
上世纪80年代中期,威斯康星大学麦迪逊分校Oliver Smithies、犹他大学的Mario Capecchi分别成功利用同源重组原理改变小鼠细胞基因组中特定区域,为基因敲除技术的发展奠定基础。届时,英国卡迪夫大学的Martin Evans开发出培育小鼠胚胎干细胞的方法,为验证基因打靶技术提供了细胞模型。1987年,Capecchi和Smithies团队利用同源重组和目标载体成功编辑小鼠胚胎干细胞的基因。
随着PCR和基因测序技术的发展,对小鼠基因组的读取越来越便捷,但是同源重组是利用载体将外源基因导入受体细胞染色体上,通过交换达到编辑基因的目的,其编辑效率极低。
1991年,约翰霍普金斯大学的Nikola Pavletich和Carl Pabo团队发现了锌指重复结构,每个锌指结构能够识别3个碱基。1994年,纽约斯隆凯特林研究所的发育生物学家Maria Jasin团队筛选到一种核酸内切酶,能够增加同源重组的概率。2001年,研究人员成功将改造的锌指蛋白和核酸酶结合,从而靶向切除非洲爪蟾蜍细胞特定的DNA片段。
以上研究奠定了锌指核酸酶(Zinc-finger nuclease, ZFN)技术的发展。ZFN由锌指DNA结合域和限制性内切酶的DNA切割域融合而成。ZFN技术的原理是通过人为改造锌指DNA结合域,靶向定位于不同的DNA序列,从而使得ZFN可以结合复杂基因组中的目的序列,并由DNA切割域进行特异性切割。
杜克大学的分子生物学家Charles Gersbach团队开发出转录激活样效应因子核酸酶(transcription activator-like effector nuclease, TALEN)系统。与锌指蛋白类似,TAL效应因子(TALE)同样具有特异性结合能力,将其与FokI核酸酶结合,即构成具有特异性基因编辑功能的武器——TALEN。TALEN技术曾被科学杂志列入十大科技突破行列(2012年)。
通过诱导DNA双链断裂(DNA double-strand break)来刺激容易出错的非同源末端连接或在特定基因所在的位置进行的同源定向修复,TALEN和ZFN能够完成一系列遗传学编辑修饰操作。
基因编辑领域的发展快速远超我们的想象,尤其是CRISPR/Cas技术。作为最新成形的基因编辑工具,CRISPR技术能够完成RNA导向的DNA识别及编辑,为构建更高效的基因定点修饰技术提供了全新的平台。2012年,Pubmed录入相关文献仅为126篇,2016年围绕CRISPR发表的最新研究数量已经增长了10倍。
当然,每一种技术都有其局限性,这也是科学家始终在寻找更为优化的基因编辑工具的根源。这些技术如何造福人类,还需要投入很多工作和时间。
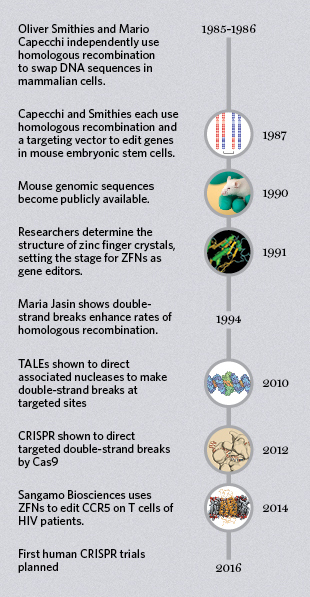
1987: Emw/Wikimedia Commons; 1990: iStock.com/sidsnapper; 1991: NCBI; 2010: David Goodsell/Wikimedia Commons; 2012: molekuul_be/Shutterstock.com; 2014: Thomas Splettstoesser/Wikimedia Commons
神经科学

纽约大学神经科学研究所的神经科学家György Buzsáki早在上世纪80年代就试图进入小鼠大脑。当时尚在加州大学圣地亚哥分校工作的他通过乙醚、低温麻醉小鼠,利用颅骨钻孔将镀金不锈钢电极植入小鼠大脑。借助这些电极小板,他可以监测到小鼠运动、认知、记忆过程中,其大脑单个神经元的电压变化。
当时最多只能放置16个电极设备,而现在,科学家们可以借助硅电极芯片同时测量1000个神经元的变化。利用探针测量大脑电生理学变化或者脑电图(EGG)测试依然是神经成像实验室的主流技术。
神经科学领域另一个发展了几十年的传统方法是膜片钳技术(patch clamping),发展自上世纪70年代末期。膜片钳技术利用离子通道的电流反映细胞膜单一或多个离子通道的分子活动。然而,膜片钳技术是侵入性的,且因为会干扰细胞正常功能而不能长时间使用。
所以,科学家一直在寻找优化的方法。随着传感器和显微技术的发展,功能钙成像技术越来越受到欢迎,其原理是通过荧光染料信号的改变反映细胞内游离钙离子浓度,以此代表细胞的功能状态。
1977年,科学家首次将磁共振成像(MRI)运用于人身上,可以无创性的获取大脑结构图。磁共振成像利用外磁场和物体的相互作用实现成像,对大脑无反射性危害。但是直至功能性磁共振成像(fMRI)的出现,才真正推进了MRI在神经影像学领域的广泛应用。fMRI利用磁振造影来测量神经元活动所引发之血液动力的改变。
除了成像,操控大脑也是科学家们试图解析大脑工作机理的另一途径。2005年,利用光线操控神经元的技术出现,由此打开了光遗传学的应用前景。MIT的Edward Boyden和斯坦福大学的Karl Deisseroth及其同事将响应光离子的通道植入哺乳动物神经细胞。利用光遗传学技术,科学家们可以改变或者换刺激小鼠大脑内的特定神经元,从而关闭癫痫发作、抑制激进行为。2007年,除了光学,科学家们试图利用化学物质实现控制的目的,他们利用G蛋白偶联受体的存在,通过注入合成的特异配体刺激或者沉默神经元。
需要清醒的是,即便科学家们现在已经找到同时记录神经元活动以及操控啮齿动物大脑细胞的技术,但是人类大脑依然是一个谜。
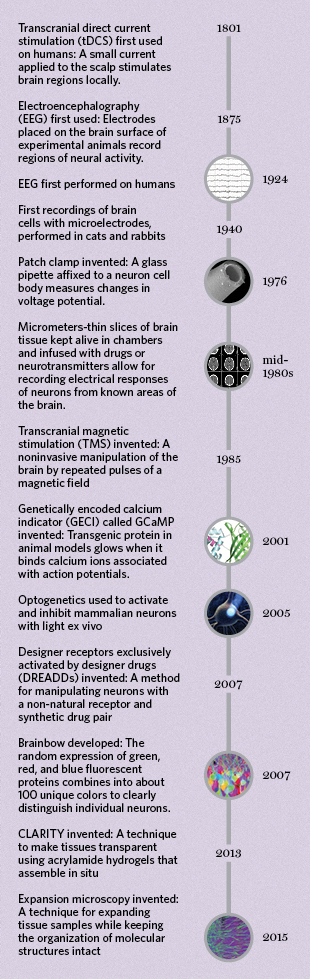
1924: Andrii Cherninskyi/Wikimedia Commons; 1976: N. Fertig/Wikimedia Commons; 1980’s: Radiology, Uppsala University Hospital; 2001: Akerboom, Rivera, Guilbe, Malavé, Hernandez, Tian, Hires, Marvin, Looger, Schreiter ER/Wikimedia Commons; 2005: MIT MCGOVERN INSTITUTE, JULIE PRYOR, CHARLES JENNINGS, SPUTNIK ANIMATION, ED BOYDEN; 2007: Jeff W. Lichtman and Joshua R. Sanes/Wikimedia Commons; 2015: Ed Boyden, Fei Chen, Paul Tillberg/MIT
显微镜

显微镜是由一个透镜或几个透镜的组合构成的一种光学仪器,是人类进入原子时代的标志。从共焦荧光显微镜到超分辨率、实时三维成像,该领域也经历着日新月异的变革。
第一台显微镜诞生于17世纪荷兰,其透镜分辨率达到1微米,由A•V•Leeuwenhoek(列文虎克)打造。1873年,物理学家Ernst Karl Abbe提出显微镜的分辨率受限于光的衍射,且传统的光学显微镜分辨率有一个物理极限,即所用光波波长的一半(0.2微米)。同时,他与Carl Zeiss、Otto Schott合作研发出分辨率达到0.2微米的显微镜。
然而,随后的发展却打破了这一预言。2000年,Stefan Hell试图突破分辨极限,成功发明出受激发射损耗荧光成像技术(STED),能够高分辨率扫描整个样品。2004年,Eric Betzig基于光激活原理(William Moerner发现),研发出光激活定位显微镜(PALM)。
分辨率的问题得到解决之后,科学家开始思考另一个问题:如何避免对样本的损害。Betzig为此对STED进行了改造,降低光水平。此外,结构光学显微镜(SIM)通过较为“温和”的光激活实现成像。2004年,欧洲生物学实验室(EMBL)的物理学家Ernst Stelzer团队研发出选择性平面照明显微镜(SPIM),它能够在亚细胞水平对活体样本实现多角度观察。得益于这些技术的更新,生物学家们能够看到和探索不一样的细微世界。
2014年, Betzig、Moerner和Hell 3位科学家共同获得诺贝尔化学奖,以表彰他们在超分辨率荧光显微镜领域做出的贡献。

1673: Wellcome Images; 1886: DrBob/Wikimedia Commons; 1987: Ernst Stelzer; 2000: Ernst Stelzer; 2004: Philippe Girard; 2006: ZEISS Microscopy; 2014: Betzig Lab, HHMI
备注:文章编译、整理自The Scientist网站“Thirty Years of Progress”,图片皆来源于特刊。