人基因组上大约有28,000个基因,其中95%的基因转录得到的mRNA前体序列会经过可变剪切的方式产生多个isoform ,也就是我们在转录组中分析经常会提到的“转录本”。可变剪切的存在,使得基因在功能调控和蛋白多样性上有较高的灵活性,同时,也会产生一些促进癌症细胞生存、增殖以及新陈代谢的剪切变体。
在过去的几十年研究当中,已经证实可变剪切参与到了例如细胞增殖、细胞凋亡、组织缺氧、血管生成、免疫逃逸和新陈代谢等一系列致癌过程当中。这些与癌症形成相关的可变剪切事件,不仅仅是基因调控紊乱的后果,而且也是癌症后期发展的激活因素。
举例说明
调控细胞凋亡的Bcl-2家族基因,其编码的不同转录本既有抗凋亡又有促凋亡的功能。
丙酮酸激酶基因 (PKM) ,其编码的转录本PKM2在肿瘤细胞中的表达显著高于正常组织,对癌细胞的代谢和肿瘤组织生长的影响不容忽视。
随着高通量测序成本的逐渐降低,越来越多的测序技术应用到了临床分析当中。作为目前最大的癌症基因信息的数据库,TCGA已经收录了超过11,000个的34种不同癌症类型病患的RNA-seq测序数据,以及其生存时间、肿瘤期、组织亚型等信息。
长期以来,学者们仅仅从DNA水平的拷贝数变异、甲基化修饰和基因、miRNA表达水平的变化上来进行癌症患者的生存分析,大多数癌症转录组分析也只是通过比较癌症样本和正常组织样本,而着眼于具有癌症特异性的可变剪切模式分析,鲜有研究通过基因不同isoform的比例差别来进行生存分析,达到外显子水平的分辨率。
这项工作难点有二:
一是生存分析中常见的问题,即因截尾数据(由于失访、死亡、未愈等,在规定的结束时间内没有出现结果事件的数据)的存在导致部分数据点的缺失,从而使得剪切变体比例和患者生存时间合理模型的建立困难;
二是转录本水平定量的不确定性。
SURVIV (Survival analysis of mRNA Isoform Variation) 的提出很好地解决了上述2个问题。
偷偷给告诉大家,相较于其他传统方法,SURVIV方法在测序深度为中度的测序样本中精确度更高哦!
首先,用SURVIV建立比例风险模型,估计每个外显子在每个病患中的外显子包含水平Ψk与风险率λk(t)的关系,λ0(t)为基线风险值,由全部患者的生存数据估算,病患的生存时间函数Sk(t)通过λk(t)计算得到。对每个外显子而言,外显子包含水平越高,风险概率越低,亦即存活率越高。
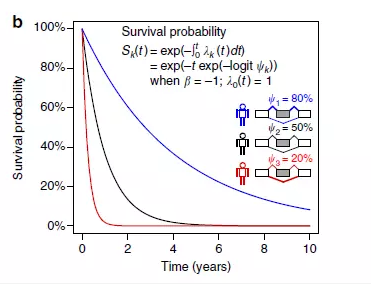
其次,考虑到样本测序深度不一的影响,SURVIV会对每个外显子在单个病患中的外显子包含水平进行不确定性估算。
案例分享
作者用SURVIV对TCGA数据库中682个浸润性导管癌(最常见的乳腺癌之一)患者的RNA-seq数据进行了生存分析。根据Croce等人的标准,将这682人的RNA-seq数据根据相应的临床参数分为26个样本含量大于50人的亚群,之后使用SURVIV方法鉴定到了229个至少在2个亚群中出现的与生存时间相关的外显子(p-value<=0.01)。
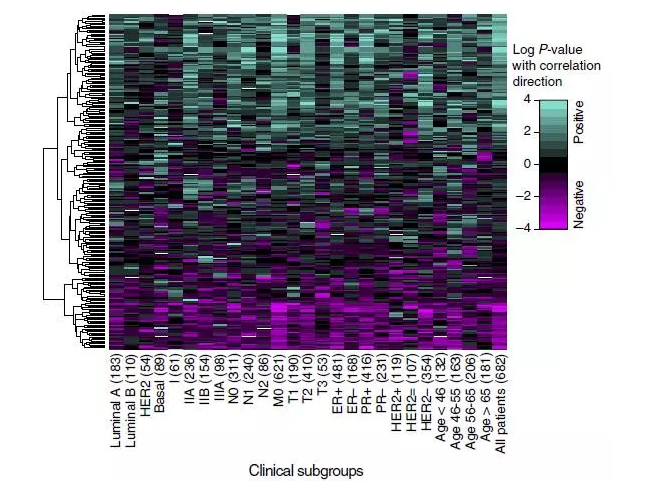
后续功能分析则发现这些外显子所在的基因功能主要与癌症相关,例如胞内信号传导、细胞凋亡、氧化应激以及DNA损伤应激。
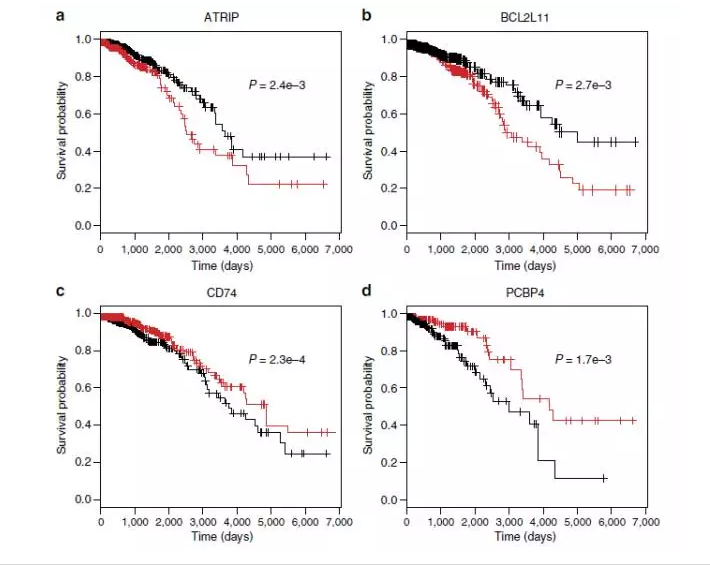
将通过SURVIV方法计算得到的229个外显子在682个癌患中的外显子包含水平进行聚类分析,绘制Kaplan–Meier生存曲线,结果表示数据能够很好的被分成2组,而这2组的生存时间确实存在较为明显的差异。
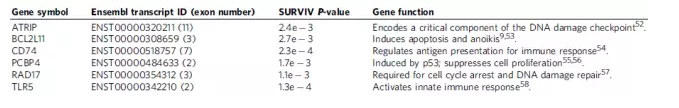
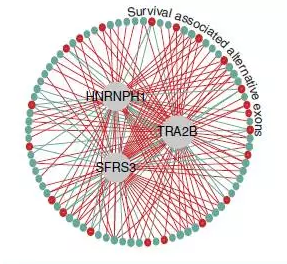
作者后续用该样本的RNA-seq数据计算了基因水平的表达量变化,得到了6个与生存时间相关的剪切因子蛋白。为了验证前述鉴定到的229个外显子剪切是否受到这些剪切蛋白的调控,构建了gene-exon 的共表达网络,发现每一个蛋白都与41~61个外显子有显著相关关系。这6个蛋白之中,TRA2B, HNRNPH1 和 SFRS3 处于调控网络的核心,与37%的外显子呈正相关或负相关的关系。
之前的研究中就已经证实这些剪切蛋白,在癌症样本中的表达水平高于正常样本,并参与到了乳腺癌的增生调节过程和很多癌症相关基因的剪切调控过程。结果表明利用SURVIV鉴定到的这些剪切事件在癌症样本中确实受到不同程度的剪切蛋白调控。
文章还将SURVIV方法与现有的、依赖于临床指标或基因水平表达变化来进行生存分析的其他方法交叉比较,验证该方法的可靠性。结果证实,依据于可变剪切分析的预测方法具有更高的准确性;使用临床指标、基因水平变化以及可变剪切多种方法的结合,在临床推广方向具有广阔的应用价值。








