T细胞受体(T cell receptor,TCR)是T细胞表面特异性识别抗原和介导免疫应答的分子,是人类基因组中多态性最高的区域之一,决定着人的免疫系统如何适应环境的变化。
T细胞受体库的多样性(包括基因重组以及选择性表达)直接反映了机体免疫应答的状态。正常个体在无抗原刺激时,TCR基因重排是随机的,因此正常人外周T细胞呈多家族、多克隆性特点。不同抗原(肿瘤、疫苗、病院微生物或者移植物等)刺激后,TCR 基因可对该抗原产生特异性识别,并使带有这类基因的T细胞得到优势扩增。
◆ 技术特点:
·多重扩增:使用多重PCR引物对TCRβ的CDR3区域进行扩增;
·高通量:一次测序得到百万级别条数的序列信息;
·高准确性:从几个到几十万个拷贝的精确计数;
·高分辨率:可以精确检测至单碱基差异;
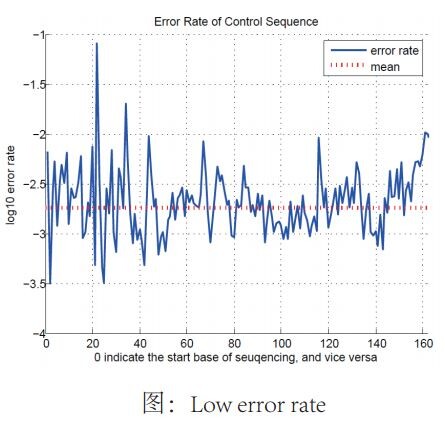
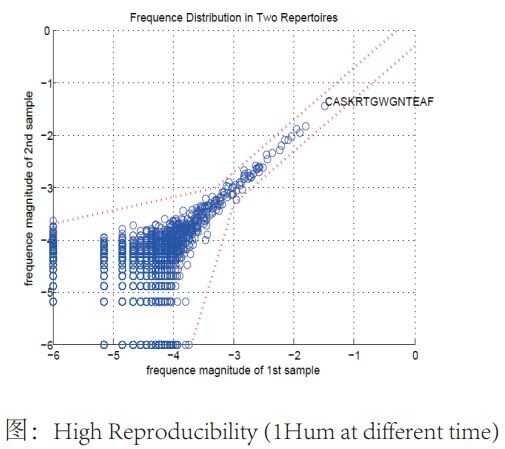
·重复性好:深度测序保证了检测的随机性,可不需技术重复;
◆ 检测流程:
TCR多样性分析:多个引物,多重PCR;
human TCR——多个 V-seg fwd primers;一个 C-seg rev consensus primer;
实验流程(引物设计,多重PCR):

mouse TCR——多个 V-seg fwd primers;一个C-seg rev consensus primer;
实验流程(引物设计,多重PCR):
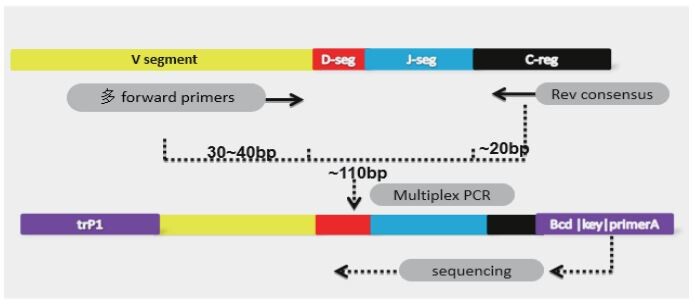

从样品中分离获得T/B细胞,之后富集TCR序列,进行高通量测序,结合生物信息学分析获得TCR重组的信息。 具体:在V基因上设计多个上游引物,在C基因上设计多个下游引物,直接进行多重扩增获取TCR的序列 B,通过5’RACE获得TCR的序列。
◆ 结果提交:
信息学分析,1. 去除污染序列;2. V, D, J, C mapping;3. CDRs 拼接碱基序列组成;
4. Vβ所属亚家族;5. V-J 组合分布 (2-D, 3-D map);6. CDR3长度分布;
7. V-J组合频率、CDR3表达频率、多态性和长度分布;8. 对缺失的碱基、V-D、
D-J插入的碱基进行分析和统计,计算其频率分布;
启因生物针对T细胞受体测序(TCR seq),采用多重扩增的手段获取TCRβ的CDR3区域,结合高通量测序技术以及生物信息学分析手段,分析VDJ重排的方式等,此技术广泛应用于肿瘤免疫、自身免疫性疾病、器官移植免疫监测、疫苗注射免疫监测等领域。
◆ 检测样品:
T细胞和B细胞,外周血单核细胞 (PBMCs),全血,组织 (包括FFPE组织),gDNA或cDNA;
◆ 检测项目:
1,人 2,鼠
T-cell receptor beta (TCRB) T-cell receptor beta (TCRB)
T-cell receptor alpha/delta (TCRA/D)
T-cell receptor gamma (TCRG)
IGH
IG kappa/lambda (IGΚ/L)
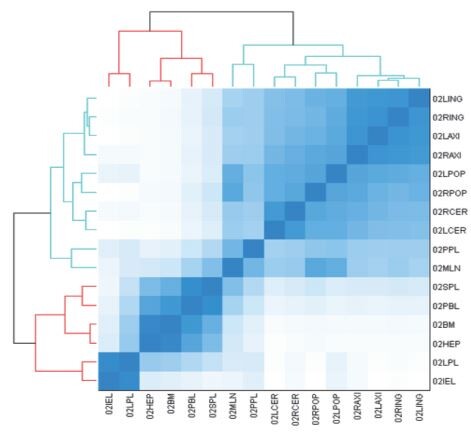
◆ 数据分析特点:
◆ 应用实例:
[1].Profiling the T-cell receptor beta-chain repertoire by massively parallel sequencing,2009,
genome Research.(见下图)
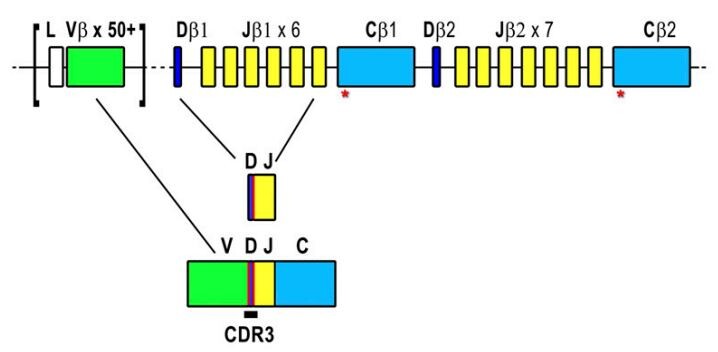
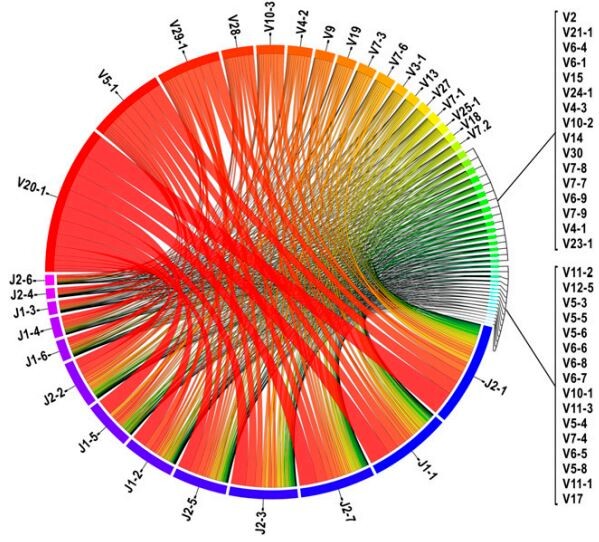
图:A,Representation of the TCRblocus at human chromosome 7q34;B,Frequencies of V-J pairing calculated from the subset of clonotypes with an unambiguousTRBVgene segment assignment (n=22,704);
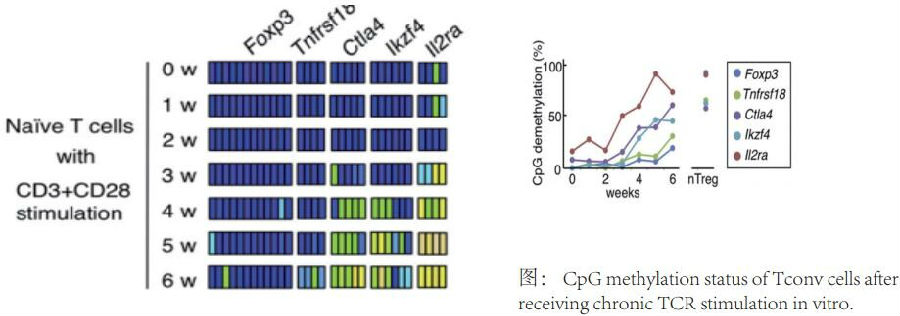
[2].T Cell Receptor Stimulation-Induced Epigenetic Changes and Foxp3 Expression Are Independent and Complementary Events Required for Treg Cell Development,2012,Immunity.(见下图)
[3].A role for cohesin in T-cell-receptor rearrangement and thymocyte differentiation,2011,Nature.
[4].Linking T-cell receptor sequence to functional phenotype atthe single-cell level,2014,Nature
Biotechnology.







