使用RNA-seq手段对实验样本进行转录组分析,关注个体或者组织器官在不同环境条件下基因表达的动态变化,挖掘生物对逆境适应的分子机制。
方案设计思路建议:
(1)植物个体受到较多环境因素的影响,包括温度、干旱、涝害、光照、盐碱、污染物、虫害以及病原菌侵染等等,相应采取的应答策略也较为多样,可以通过激素信号分子和细胞表面受体调控相关基因的表达。建议设置多个关键处理时间点,研究植物个体短时间内和长时间内转录组动态变化趋势;揭示同一器官组织在不同环境胁迫下或不同器官组织在同一环境胁迫下基因的时空表达模式;
(2)动物器官组织样本主要来源畜牧动物、水产动物、昆虫、人以及模式动物小鼠和线虫,关注点为温度处理、光照处理、污染物处理、病原菌/虫感染、用药前后特异性表达基因,根据基因表达模式分析揭示器官组织响应环境胁迫过程的分子机制;
(3)对于微生物样本,主要通过环境转录组分析不同培养环境、次生代谢产物积累以及毒素产生差异基因表达,解析环境诱导活性物质生产的分子机理。
表:环境转录组取材策略建议
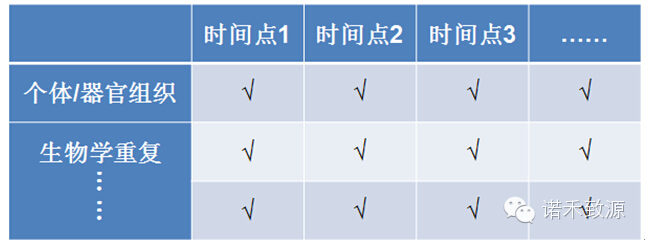
要点说明:
a. 基于时间递进轴,分析相邻时间点的差异基因,组成聚类分析的基因集合。
b. 至少需要2次生物学重复,3次以上的生物学重复更好。已有研究表明生物学差异是基因自身表达的特性,与检测技术的选择以及数据处理的方式无关[1,2]。
c. 对于基因组已发表的物种,以基因组为RNA-seq基因定量的参考序列;对于基因组尚未发表的物种,使用Trinity 软件对所有样本的测序序列进行混合拼接后得到转录组参考序列[3,4],作为基因定量的参考序列。

参考文献:
[1] Hansen K. D., et al.,Sequencing technology does not eliminate biological variability. NatureBiotechnology, 2012
[2] Trapnell C., et al., Differential analysis of gene regulation attranscript resolution with RNA-seq. Nature Biotechnology, 2013
[3] Grabherr M. G., et al., Full-lengthtranscriptome assembly from RNA-Seq data without a reference genome. NatureBiotechnology, 2011
[4] Duan J., et al., Optimizing de novo common wheat transcriptome assemblyusing short-read RNA-Seq data. BMC Genimics, 2012
敬请期待案例解析!







