GWAS研究搜索整个基因组,查找单个DNA变化与生物体属性(如抗生素耐药性)有关的位点。要出现这种情况,就必须发生一个称为重组的遗传交换过程。这是两段DNA序列结合、交换遗传数据和重排单一变化组合的部位。由于重组(在人类中是很常见的)在细菌中很罕见,所以在这之前,研究人员无法定位构成一个基因的碱基序列中的单个变化。直到现在,才有可能定位一般区域,在这些区域中,变化发生在所谓的镶嵌基因(包含来自多个菌株的遗传学数据)中。
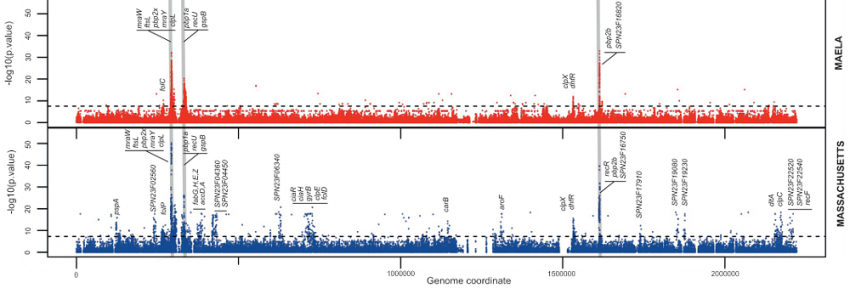
为了克服这个障碍,本文中的研究人员使用最丰富的肺炎链球菌可用数据集,由玛希隆牛津热带医学研究部门的儿科医生Claudia Turner博士和临床微生物学家Paul Turner博士收集。研究人员从缅甸和泰国边境一个难民营的近1000名婴儿和母亲中,分离出3000多种肺炎链球菌的样本数据集,可提供足够的重组事件,为研究人员提供了关于引起耐药性的位点变化的精确数据。
第二组隔离菌群(几百个)是从马萨诸塞州收集,作为项目的一部分,旨在评估疫苗对2001年引入美国的肺炎链球菌的影响。
本文资深作者Stephen Bentley教授称:“在这项研究中,我们已经表明,这种强大的遗传工具,改变了我们对于人类遗传学的理解,可以应用于细菌。这为抗生素耐药性、传播和毒性研究,开辟了新的途径,而以前在细菌基因组学中研究这些方面,被认为是不可能的。”
该研究的下一阶段将包括,调整这项技术,以能够识别细菌中那些使菌株更致命的基因,和那些使菌株在寄主之间传播的基因。随着基因测序进入诊所,这种详细的理解,将会让我们找到更好的控制和治疗策略。
Julian Parkhill教授说:“揭开耐药性背后的所有单字母差异,对于未来用基因组测序预测临床微生物的抗生素敏感性,是至关重要的。GWAS,可让我们能够找出真正的遗传原因,从而能够做到这一点。”







