上篇:表达谱芯片质的飞跃:人类全转录组芯片HTA2.0(一) (https://https://www.biodiscover.com/news/research/110454.html?key=1073bcab17c99f0f)
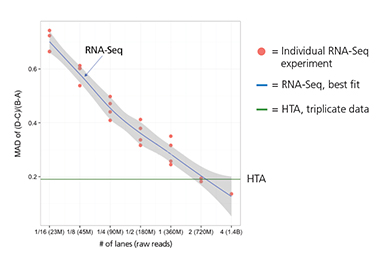
RNA-Seq的准确性取决于测序深度。HTA芯片提供了准确的结果,其准确性相当于2个Lane的RNA-Seq数据。如下图通过一个线性样本混合物模型(Linear Tissue Mixture Model)评估了所有外显子的表达水平检测的准确性,该模型根据预先设定的比例混合两个RNA标准品。y轴是检测错误或(D-C)/(B-A)的MAD (Mean Absolute Deviation,平均绝对偏差)的绝对值,其中标准品A=Universal Human Reference RNA (UHRR),Agilent Technologies,Inc.;标准品B=Human Brain Reference RNA (HBRR),Life Technologies,Inc.;C=75%A+25%B;D=25%A+75%B。HTA芯片提供的全转录组基因表达测定的准确性相当于HiSeq™+2000系统上2个Lane测序数据所带来的准确性。
*Let Professionals Serve Professionals基因有限公司 www.genecompany.com*
方便快捷的数据分析
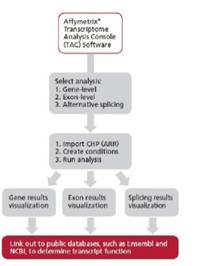
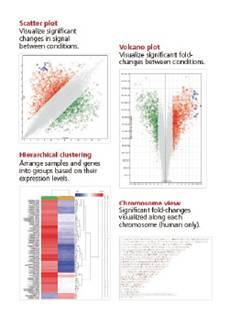
RNA-SEQ往往花费太长的时间从庞大的测序结果中获得有意义的数据,这个过程需要研究人员在信息学上投入大量的时间和资源。而HTA芯片可以在一周之内完成从RNA到生物学结果的转换,整个流程可以支持少量或者大量的样本研究;基于为研究人员设计的简单易用的免费数据分析软件Transcriptome Analysis Console (TAC) Software,研究人员可以简便操作,更易获得直观的数据。TAC软件可以帮助研究人员进行基因水平、外显子水平的表达量分析和可变剪接分析, 操作简便,只需四步即可进行完整的生物信息分析;可以形象化的结果展示工具,能够直观的展示芯片中发现的重要差异基因;能够发现、区分并展示不同 transcript isoforms;能够将结果与公用数据库链接比对。
•如下图是TAC的分析流程以及部分分析结果示意图。
来自终端用户的好评
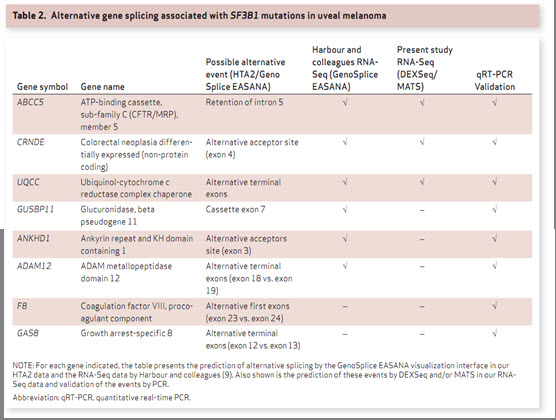
HTA芯片于2013年面世,就受到研究人员的追捧,在实践中历经考验。以下是英国曼彻斯特癌症研究所发表的内容,在他们的研究中,关注了SF3B1基因突变对转录组引发的可变剪接变化。他们分别使用了RNA-SEQ和HTA2对可变剪接事件进行预测,并用RT-PCR对可变剪接事件进行验证。HTA2预测出8个可变剪接事件,而2个独立的RNA-SEQ分别只预测到其中的5个和3个事件。而后续的验证实验表明,HTA2预测出的8个可变剪接事件都是准确的。
原文链 https://cancerdiscovery.aacrjournals.org/lookup/doi/10.1158/2159-8290.CD-13-0330。