2类V型CRISPR效应物Cas12被认为是从转座子相关TnpB蛋白的IS200/IS605超家族进化而来的。最近的研究已经确定TnpB蛋白是一种微型RNA引导的DNA内切酶。TnpB与单个长RNA (ωRNA)结合,并切割与ωRNA向导互补的双链DNA靶标。然而,TnpB在RNA引导下的DNA切割机制及其与Cas12酶的进化关系尚不清楚。
2023年4月5日,博德研究所张锋及东京大学Osamu Nureki等团队合作在Nature 在线发表题为“Cryo-EM structure of the transposon-associated TnpB enzyme”的研究论文,该研究报道了耐辐射球菌(Deinococcus radiodurans)ISDra2 TnpB及其同源ωRNA和靶DNA复合物的冷冻电镜结构。
在结构上ωRNA采用了一种意想不到的结构,形成了一个假结,在所有Cas12酶的引导RNA中是保守的。此外,该结构以及功能分析揭示了紧凑的TnpB如何识别ωRNA并切割与向导互补的目标DNA。TnpB与Cas12酶的结构比较表明,通过不对称二聚体形成或多种REC2插入,CRISPR-Cas12效应物获得了识别引导RNA-靶DNA异源双链体的原间隔-邻近基序-远端的能力,从而参与CRISPR-Cas适应性免疫。总的来说,该研究提供了TnpB功能的机制见解,并推进了我们对从转座子编码的TnpB蛋白到CRISPR-Cas12效应物的进化的理解。
另外,2023年6月28日,博德研究所张锋团队在Nature 在线发表题为“Fanzor is a eukaryotic programmable RNA-guided endonuclease”的研究论文,该研究报告了Fz的生化特性,表明它是一种RNA引导的DNA内切酶。该研究还表明Fz可以被重新编程用于人类基因组工程应用。该研究利用冷冻电镜分析了Spizellomyces punctatus Fz (SpuFz)在2.7Å的结构,揭示了Fz、TnpB和Cas12之间的核心区域的保守性,尽管同源RNA结构不同。总之,该研究结果表明Fz是真核生物的OMEGA系统,表明RNA引导的内切酶存在于生命的所有三个领域。
2023年4月6日,博德研究所张锋团队在Science 在线发表题为“Structure of the R2 non-LTR retrotransposon initiating target-primed reverse transcription”的研究论文,该研究报道了Bombyx mori R2 non-LTR逆转录转座子在其核糖体DNA靶点处启动TPRT的冷冻电子显微镜结构。目标DNA序列在插入位点被解开,并被上游基序识别。逆转录酶(RT)结构域的延伸识别逆转录转座子RNA,并引导3 '端进入RT活性位点以模板逆转录。该研究使用Cas9在体外将R2重定向到非原生序列,这表明未来可以作为一种基于可重编程RNA的基因插入工具。
2023年3月29日,博德研究所张锋团队在Nature 在线发表题为“Programmable protein delivery with a bacterial contractile injection system”的研究论文,该研究发现Photorhabdus毒力盒(PVC)——来自昆虫病原细菌Photorhabdus asymbiotica的eCIS——的目标选择是由PVC尾纤维的远端结合元件对目标受体的特异性识别介导的。在尾巴纤维的结构引导工程中,该研究表明PVC可以被重新编程,以靶向这些系统不天然靶向的生物(包括人类细胞和小鼠),效率接近100%。最后,该研究发现PVC可以装载不同的蛋白质载荷,包括Cas9、碱基编辑器和毒素,并可以将它们功能性地输送到人类细胞中。总之,该研究结果表明PVC是一种可编程的蛋白质传递装置,可能应用于基因治疗、癌症治疗和生物控制。
2023年1月5日,博德研究所/哈佛医学院/麻省理工学院张锋团队在Cell在线发表题为“A transcription factor atlas of directed differentiation”的研究论文,为了全面了解TF及其控制的程序,该研究创建了一个包含所有注释的人类TF剪接异构体的条形码库(> 3500),并将其应用于构建TF图谱,以单细胞分辨率绘制过表达每个TF的人类胚胎干细胞(hESCs)的表达谱。该研究将TF诱导的表达谱映射到参考细胞类型,并验证候选TF用于生成不同的细胞类型,涵盖所有三个胚层和滋养层。具有库子集的靶向筛选使研究人员能够创建定制的细胞疾病模型,并整合mRNA表达和染色质可及性数据,以识别下游调控因子。最后,该研究通过开发和验证一种预测TF组合的策略来描述组合TF过表达的影响,该策略产生匹配参考细胞类型的目标表达谱,以加速细胞工程工作。

原核生物中的CRISPR-Cas系统提供针对外源核酸的适应性免疫,分为两类(1类和2类)和六种型(I-VI型)。2类系统包括II型、V型和VI型,其中Cas9、Cas12和Cas13分别是负责干扰的效应酶。II型Cas9效应蛋白与双RNA向导(CRISPR RNA (crRNA)和反式激活crRNA (tracrRNA)或其人工连接的单向导RNA (sgRNA))结合,并利用HNH和RuvC核酸酶结构域切割双链DNA (dsDNA)靶点。V型Cas12效应蛋白进一步分为Cas12a-m亚型,具有不同的功能。
虽然Cas12蛋白通常有一个单一的RuvC核酸酶结构域,但它们在这个保守区域之外的序列相似性很低。Cas12酶与crRNA向导或双RNA向导(crRNA和tracrRNA)结合,利用其单一RuvC结构域切割dsDNA靶标。最近的报道表明,2类CRISPR效应子Cas9和Cas12分别独立于IS200/IS605转座子编码核酸酶超家族的两个成员IscB和TnpB进化。IscB的功能和结构研究表明,相关的ωRNA(专用性移动元件引导活性(OMEGA))在识别引导RNA-靶DNA异源双链体中起着至关重要的作用,使IscB能够利用HNH和RuvC结构域切割其靶DNA。
TnpB蛋白也是RNA引导的DNA内切酶。TnpB与单个长链非编码RNA (ωRNA,也称为右端元件RNA (rRNA))结合。编码ωRNA的基因与tnpB基因的3′端和转座子的非编码右端元件重叠,TnpB利用其RuvC结构域切割与ωRNA引导序列互补的dsDNA靶标。此外,TnpB需要目标序列上游的靶邻基序(TAM)来切割目标DNA,类似于Cas酶需要原间隔物邻近基序(PAM)序列来切割目标DNA。
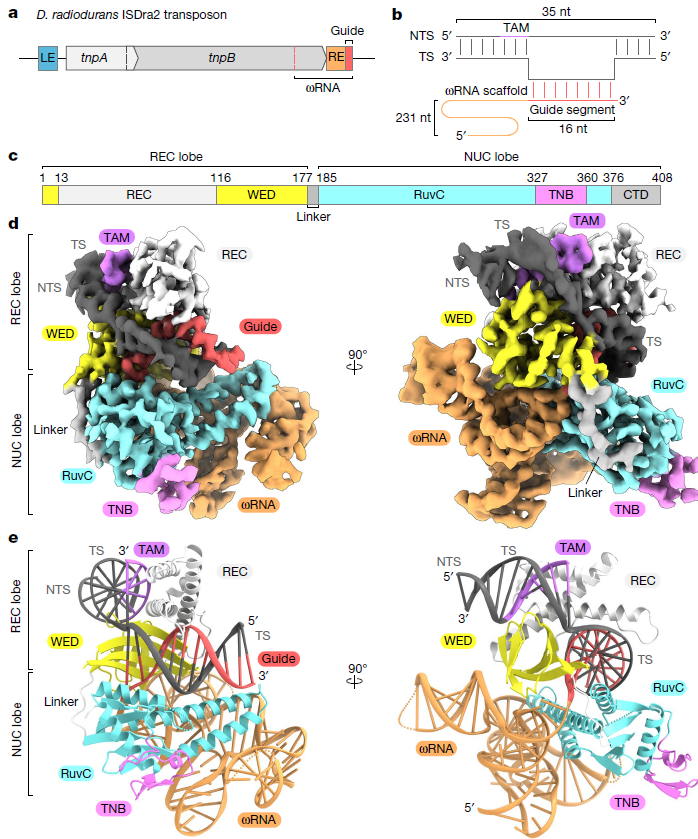
TnpB-ωRNA-靶DNA三元复合物的冷冻电镜结构(图源:Nature)
耐辐射球菌ISDra2的TnpB(为简单起见简称TnpB)由408个残基组成,结构域组织与Cas12f(V型Cas12酶中最小的亚型)相似。然而,最近的一项分子质量分析显示,TnpB作为单体发挥作用,而Cas12f作为二聚体发挥作用。此外,Cas12f与双RNA向导结合,而TnpB与单ωRNA结合。因此,TnpB蛋白如何与其同源ωRNA组装以介导RNA引导的双链DNA断裂仍然未知。
该研究报道了耐辐射球菌(Deinococcus radiodurans )ISDra2 TnpB及其同源ωRNA和靶DNA复合物的冷冻电镜结构。在结构上ωRNA采用了一种意想不到的结构,形成了一个假结,在所有Cas12酶的引导RNA中是保守的。此外,该结构以及功能分析揭示了紧凑的TnpB如何识别ωRNA并切割与向导互补的目标DNA。TnpB与Cas12酶的结构比较表明,通过不对称二聚体形成或多种REC2插入,CRISPR-Cas12效应物获得了识别引导RNA-靶DNA异源双链体的原间隔-邻近基序-远端的能力,从而参与CRISPR-Cas适应性免疫。总的来说,该研究提供了TnpB功能的机制见解,并推进了我们对从转座子编码的TnpB蛋白到CRISPR-Cas12效应物的进化的理解。
文章来源“iNature”