该研究首创在预训练模型中引入蛋白质动态时空信息,在药物-蛋白亲和力预测任务等多个下游任务的表现上大幅超越目前最优模型。该模型可辅助药物化学专家更加精准的筛选出高活性小分子,从而加速临床前研发。该研究是近年来药物-蛋白亲和力预测领域发表的影响因子最高的论文之一。

基于靶点的药物筛选是基于功能蛋白进行的亲和力或功能筛选,因此获得有足够活性的小分子是实现药物筛选成功的关键要素之一,准确预测小分子和靶蛋白之间的结合亲和力是药物开发过程中的一个重大挑战。
在此项研究中,德睿智药将团队制药经验与AI技术深度融合。基于对蛋白质结构多样性的深入理解,团队构建了具有结构代表性的蛋白质“小样本”数据集,进行蛋白质动力学模拟后,获得其动态信息,并以此为基础建立AI预训练模型。该模型表现超越以往基于蛋白质3D静态结构信息的AI模型,达到目前最优,为药物-蛋白亲和力筛选提供了强有力的工具。
国际人工智能学界权威,IEEE Fellow,西湖大学人工智能讲席教授李子青(Stan Z. Li)指出:“传统的药物蛋白结合理论基于静态蛋白构象假设,而实际上蛋白构象在药物结合之前和之后发生了改变。此研究是预测蛋白-药物结合动态的一个尝试, 在预训练模型中首次引入蛋白质时空动态信息,并设计了等变图匹配网络,以学习药物亲和力在蛋白构象几何形变条件下的变化,更准确地完成药物-蛋白亲和力预测这一核心任务,从而提升AI药物设计的有效性。这个工作为该领域研究提供了一个新的benchmark。”
1 ProtMD框架
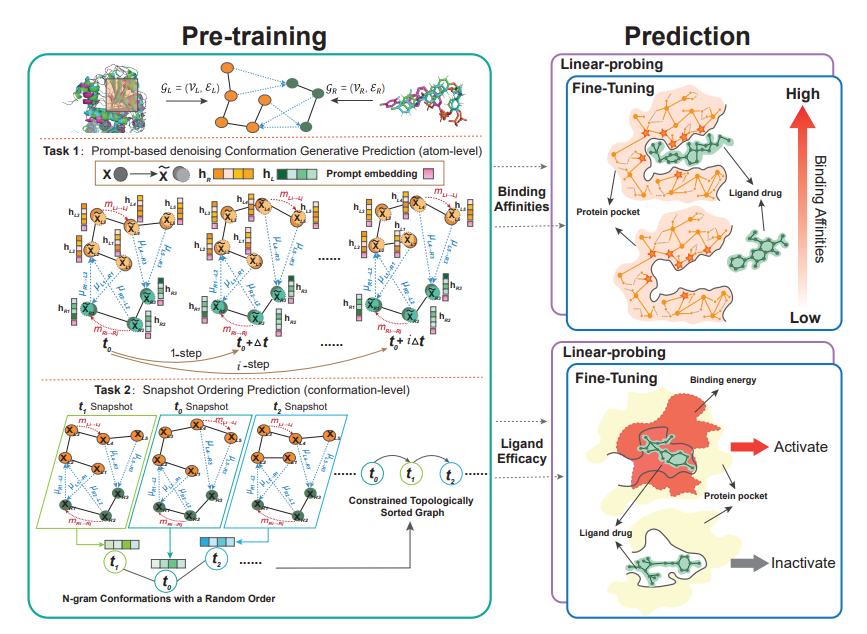
图1 ProtMD框架示意图(图源:[1])
ProtMD有两个专门设计的自监督学习任务:一个是基于Prompt的去噪生成预测,另一个是Snapshot Ordering。模型在两个下游任务上通过微调和线性探测进行验证,其中结合亲和力预测是一个回归问题,Ligand Efficacy Prediction是一个分类问题。
2 预训练任务设计
2.1 预训练数据集
为了获取无标签的蛋白质时空信息进行预训练,德睿智药团队根据先前蛋白质研究经验,从PDB蛋白质数据库共计57651个人类蛋白结构中选取了具有结构代表性的数十个蛋白质结构,使用Molecule Dance平台对这数十个蛋白质进行分子动力学模拟,获得数TB蛋白质空间动态信息用于下步预训练。
2.2 Prompt-based Denoising Conformation Generative Task
生成式自监督学习是无监督预训练的一个典型任务。为了更好地捕获构象中原子级的信息,提出了基于提示的去噪生成预测自监督任务。该任务要求模型根据当前构象产生未来构象。与朴素的生成式自监督学习不同,团队在模型中添加了一个时间序列提示Prompt来调节和控制源构象和目标构象之间的时间间隔,这使分子编码器能够捕获MD轨迹内的短期和长期依赖性,生成大于等于一步之后的构象。除此之外,额外的噪声被注入到构象中增加了该自监督生成任务的难度以防止模型过拟合。
2.3 Snapshot Ordering Prediction
此前已有大量研究证实,每种构象的形状和表面都带有了解潜在分子相互作用的关键信息。然而,只利用基于提示的去噪生成任务是建立在原子水平上的,无法有效捕获构象级全局信息。为了获取构象级别的信息,团队设计了一个快照排序任务。具体来说,通过将一组密切相关的构象排序为连贯的子轨迹,教会模型从全局角度理解它们的依赖关系。
目前预训练模型参数量越来越大,如BERT-base参数个数达1.1亿个[1],使用此类模型面临着较大的性能瓶颈。为加速ProtMD的训练及推理过程、便于后续工业级大规模使用,我们训练了两个版本的ProtMD,即仅有1万参数的轻量级版本,以及有522万参数的完整版本。
3 实验结果
3.1 药物亲和力预测
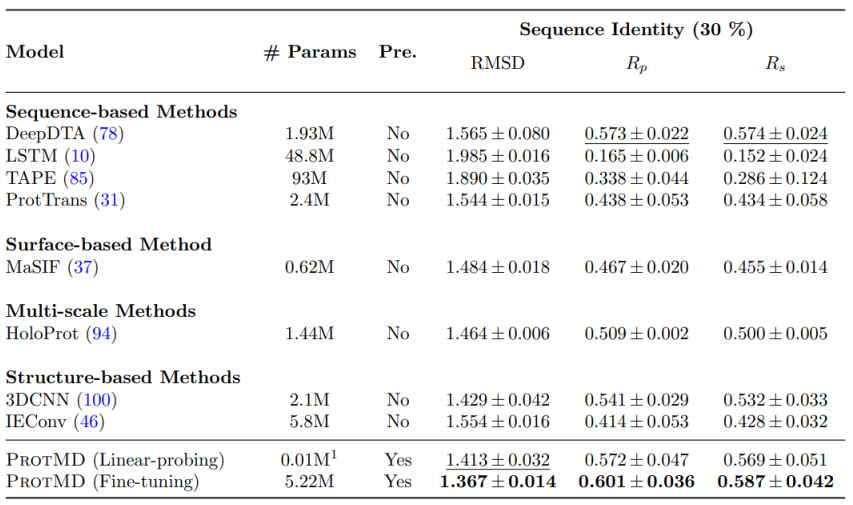
在药物-蛋白亲和力预测任务上,ProtMD仅有1万参数的轻量级版本表现已超过以往最优(SOTA)模型,在522万参数版本达到1.367的RMSD和最佳的Pearson和Spearman系数,较SOTA模型提升5%表现。轻量级版本的参数个数比以往各类模型小50倍以上,ProtMD的更优表现表明了模型的高学习效率,引入蛋白质时空动态信息可显著提升药物亲和力预测效果。
3.2 Ligand Efficacy Prediction
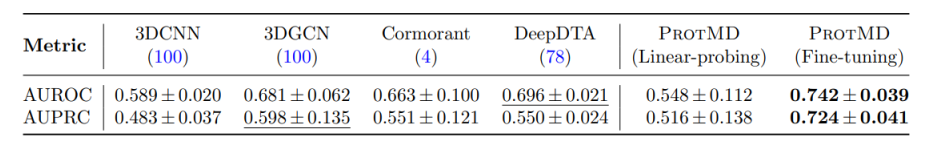
在Ligand Efficacy Prediction[2,3]任务上,ProtMD 522万个参数版本AUPRC达到0.724,较SOTA模型提升14%表现。
4 总结
ProtMD的研发为基于蛋白的机器学习预训练模型研发提供了新方向,证明了蛋白质动态时空信息在蛋白-小分子亲和力预测上的重要性。既由该底层原理上的突破,该模型预测精度达到“同类最优”。ProtMD的工业级版本可大幅提高药物亲和力预测与虚拟筛选效率。
参考资料:
[1]Devlin J, Chang M W, Lee K, et al. Bert: Pre-training of deep bidirectional transformers for language understanding[J]. arXiv preprint arXiv:1810.04805, 2018.
[2]Townshend R J L, Vögele M, Suriana P, et al. Atom3d: Tasks on molecules in three dimensions[J]. arXiv preprint arXiv:2012.04035, 2020.
[3]Friesner, R. A., Banks, J. L., et al. (2004). Journal of Medicinal Chemistry, 47(7), 1739–1749.