01 研究成果
2022年8月17日,青岛华大基因研究院、深圳华大生命科学研究院联合中国农业科学院北京畜牧兽医研究所、山东大学微生物技术国家重点实验室、丹麦哥本哈根大学等单位在Environmental Microbiome(IF6.36)上发表了题为 “Sequencing introduced false positive rare taxa lead to biased microbial community diversity, assembly, and interaction interpretation in amplicon studies” 的文章。

■ 该研究采用华大智造 DNBSEQ-G400 (MGISEQ-2000)及Illumina NovaSeq 6000两个主流测序平台,对微生物模拟群落、表层海水、红树林沉积物、小鼠肠道、牛瘤胃等不同生态系统的样本进行 16S V4 扩增子测序分析,发现NovaSeq 平台存在显著的批次效应,其由于标签跳跃(index hopping)导致的假阳性或假阴性稀有类群对微生物的群落组成、多样性、相互作用及群落构建机制等研究造成偏差。
■ 第一作者:贾洋洋、赵圣国
通讯作者:
陈建威 (chenjianwei@genomic.cn)
章文蔚 (zhangww@genomics.cn)
Karsten Kristiansen (kk@bio.ku.dk)
02 研究背景
越来越多的研究表明,低丰度的稀有类群在微生物群落中可能有着重要的生物功能和生态贡献。然而,稀有微生物类群的研究受其固有的稀缺性和现有技术不足的阻碍。过去十年最广泛使用扩增子测序中,焦磷酸测序(pyrosequencing)和Illumina测序可能产生的样本标签跳跃(index hopping)会引入样本交叉污染。尽管通过下游质量控制和聚类/降噪算法可以消除测序错误及人工合成序列的污染,但没有算法可以消除由标签跳跃引入的样本交叉污染的高质量测序数据,因此难以区分扩增子研究中的真实或假阳性稀有类群。
根据 Illumina 2017 年发布的白皮书可知对于各种 Illumina 测序平台,标签跳跃的发生率可能为 0.2~6% 甚至更高;而基于DNA纳米球滚环式复制的 DNBSEQ 测序平台标签跳跃的发生率低至 0.0001-0.0004%。不同的测序平台可能有不同的优缺点,目前很少研究评估测序产生标签跳跃如何以及在多大程度上会影响对稀有微生物类群的研究。本研究使用两个不同的主流测序平台 DNBSEQ-G400 及 NovaSeq 6000 对来自商业微生物模拟群落、实验室自制模拟群落以及具有不同复杂性的多个典型生态系统微生物群落进行了测序,以系统地分析标签跳跃如何影响我们对各类微生物群落中稀有类群的认识。此外,通过对牛瘤胃微生物群落的真实案例研究,进一步表明了标签跳跃可能导致微生物组成、群落构建机制和稀有微生物的生态作用等研究结果存在偏差。
03 实验设计
本研究首先使用已知微生物组成的商业微生物模拟群落(ZymoBIOMICS™ Mock D6305)及两个实验室自制模拟群落(4bac & 7bac)来确认不同测序平台(DNBSEQ-G400, NovaSeq 6000)测序过程中是否存在引入标签跳跃以及在不同平台上的标签跳跃发生率(图 1,Part1)。随后对来自具有不同群落复杂度的几个典型生态系统的样本进行测序,通过三次平行测序分析微生物群落检测结果的稳定性,并检验标签跳跃如何影响不同环境样本的群落多样性(图 1,Part2)。最后使用了一组牛瘤胃样本(47个)进行案例研究,通过群落多样性、构建机制、互作网络及理化参数关联等分析以及假阳性OTU PCR实验验证,评估标签跳跃产生的假阳性/假阴性结果对微生物生态作用和群落构建机制等结果的影响(图 1,Part3)。
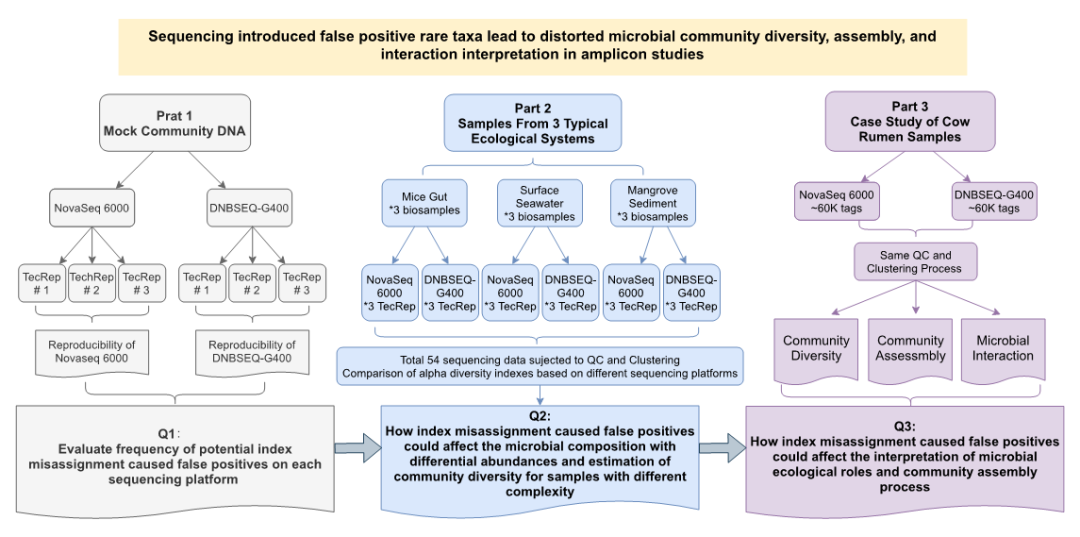
图1 本研究设计和流程示意图
04 研究成果
■ DNBSEQ-G400 平台的批次效应及假阳性率更低,NovaSeq 6000 产生的假阳性可能是标签跳跃引入的且无法通过QC去除
为了评估可能由扩增子测序中的标签跳跃引入的假阳性率,我们使用已知组成的商业微生物模拟群落(Mock D6305,含8种已知细菌)和分别包含4种已知的细菌(4bac)、7种细菌(7Bac)的两个实验室自制模拟群落对 16S V4 进行三次平行扩增测序(图 1)。对于商业Mock,DNBSEQ-G400 共获得了17个OTU(每次重复检出OTU数目分别为14、15、16个),其中3个OTU只在一次重复中出现,另外14个OTU在所有三次技术重复都有检测到。与之相比,NovaSeq 6000 共获得了162个OTU(每次重复检出OTU数目分别为92、156、66个),其中仅一次检出的OTU有67个,两次检出的38个,所有三次技术重复检出的57个。我们发现NovaSeq平台的检测结果具有显著的批次效应,与DNBSEQ的82%重复检出率相比,NovaSeq只有35%的OTU能被所有三个技术重复一致地观察到(图 2A),对4bac、7bac模拟群落的分析也揭示了相似的观察结果。
OTU的分类注释显示,Mock样本在两个测序平台所有技术重复都检测到了模拟群落的所有目标菌株,这表明在给定足够测序深度的情况下我们可以成功检测到群落中所有的高丰度微生物。然而尽管两个测序平台都成功检测到预期目标物种,但两个平台都发现了一些的与模拟群落组成不一样的可能是假阳性的 Unexpected OTU,且NovaSeq平台的 Unexpected OTU 数量几乎比 DNBSEQ 平台高出两个数量级(图 2B)。NovaSeq和DNBSEQ两个平台的 Unexpected OTU 相对丰度分别为1.19%和0.09%,占测序数据的 5.68%和0.08%。我们发现DNBSEQ平台检测到的9个 Unexpected OTUs 也均在NovaSeq平台检出,且这9个OTU中的5个可能是Mock的细菌的突变株(序列相似性97.18%~99.60%),其余4个可能是原始DNA样本中的潜在污染或分装过程中来自环境的污染,表明这9个共有的 Unexpected OTUs 更有可能来自原始测序DNA样本,而不是来自各自的测序过程(图 2B-C)。另一方面,NovaSeq的 Unexpected OTUs 物种多样性很高,且仅有小部分能在技术重复中检出(图 2B);同时在4bac、7bac的NovaSeq测序结果中也都发现了很多 Unexpected OTUs,观察结果与商业Mock类似。由于标签跳跃的发生率较低,因此我们尝试使用更严格的质量控制和更高的OTU聚类丰度阈值来消除标签跳跃产生的污染。然而,即使我们将丰度阈值提高到50也无法消除所有的潜在污染,表明标签跳跃的污染可能无法通过常规的分析方法去除。
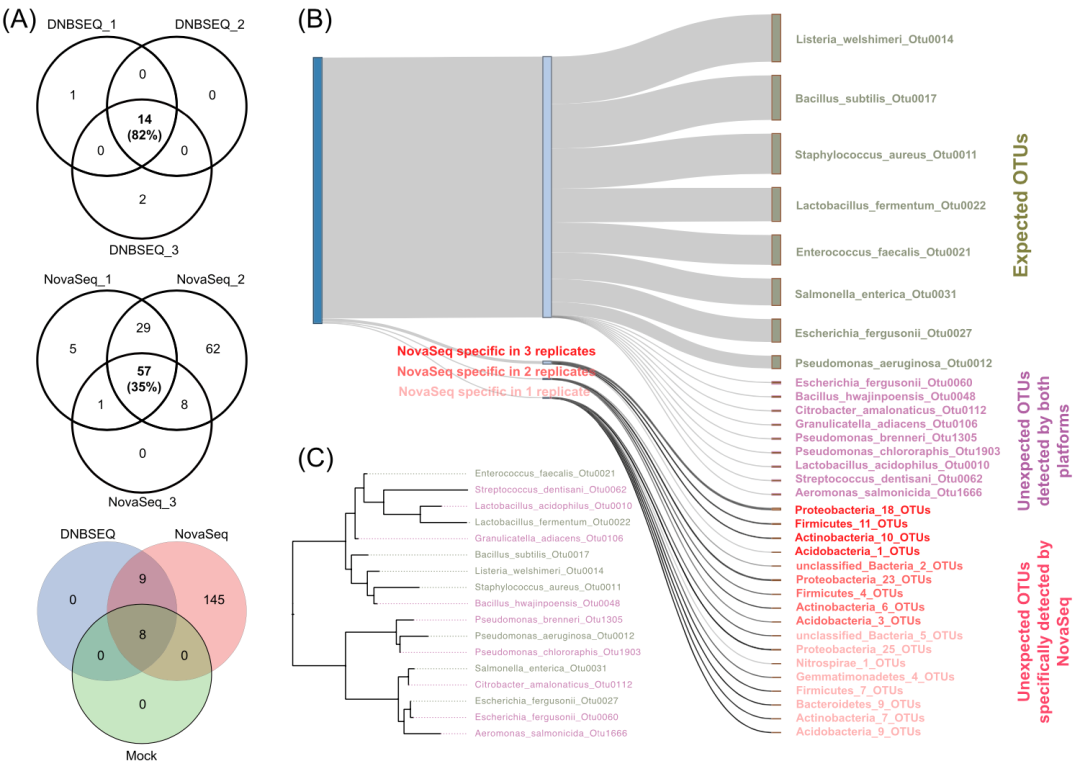
图2 商业模拟微生物群落样本(Mock)不同平台扩增子测序结果比较。
■ 稀有微生物类群更容易受到标签跳跃引物的偏差的影响
由于各类生态系统的真实微生物群落在微生物组成和丰度分布上比模拟群落复杂得多,我们推测标签跳跃可能会导致真实样本中出现更有趣的假阳性/假阴性现象。通过 DNBSEQ-G400 和 NovaSeq 6000 平台对分别来自低、中、高群落多样性的小鼠肠道、表层海水、红树林沉积物等典型微生物生态系统的样本进行了三次平行测序。对于这些来自不同微生物生态系统的真实样本,每类物种的检出率与其丰度密切相关。因此,与稀有类群相比,高丰度的丰富类群捕获测序及检出的机会要高得多。本研究中,我们将相对丰度 ≥1% 的类群定义为丰富类群(Abundant taxa, AT),<0.1% 为稀有类群(Rare taxa, RT),其余为中等类群(Moderate taxa, MT)。对每类生态系统的三次技术重复结果比较显示,DNBSEQ-G400 的技术重复可以检测到100%的丰富类群、97.53%中等类群和68.93%稀有分类群,而NovaSeq平台对应比例为100%、87.94%和39.50%(图 3A)。测序平台之间的比较显示,中等和稀有类群的平台特异类群的比例更高,特别是对于NovaSeq平台。与 DNBSEQ-G400 平台的结果相比,NovaSeq 6000 平台在海水和小鼠肠道样本中Alpha 多样性更高,但在红树林样本中显著降低(图 3B)。进一步使用红树林和小鼠肠道的宏基因组测序数据对其OTU进行比对定量,发现NovaSeq平台有更多的OTU没有被宏基因组数据检测到,表明其假阳性的风险更高。此外,Beta多样性分析的 weight & unweight UniFrac 聚类树一致表明,DNBSEQ-G400 平台三次技术重复均按其所有测试生态系统的生物样本分群,而NovaSeq的三次技术重复按测序批次或随机方式分群,表明其群落结构受测序批次影响大。
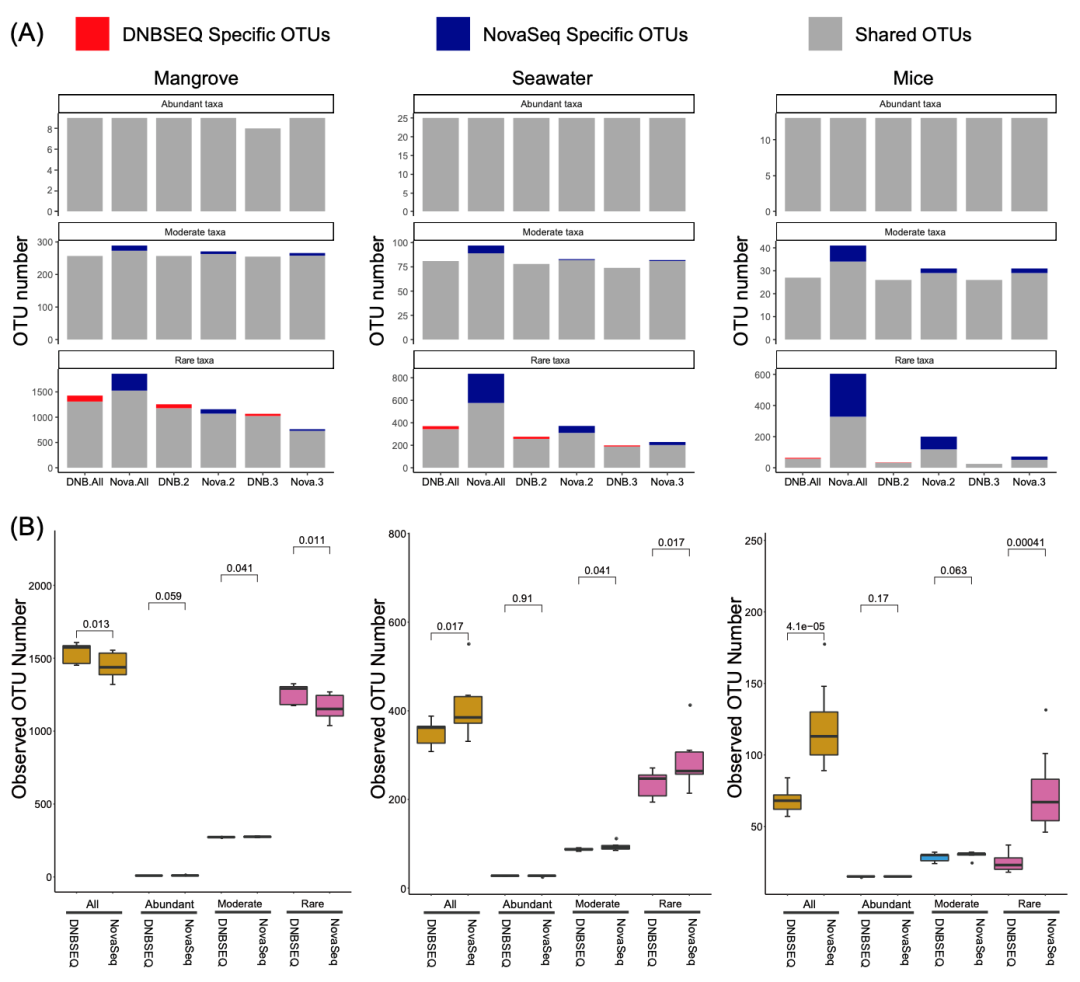
图3 三种典型生态系统样本DNBSEQ与NovaSeq扩增子测序结果比较。
■ 牛瘤胃微生物群落不同测序平台结果比较
虽然目前已经对牛瘤胃生态系统进行了大量研究以阐明微生物的组成和功能多样性,但以前的大多数研究都忽略了其中稀有类群。为了研究瘤胃稀有类群的生态特征,并评估不同测序平台稀有群落结果的差异,我们在 DNBSEQ-G400 和 NovaSeq 6000两个平台上对47个牛瘤胃液样本进行了扩增子测序并整合分析。在总共3043个OTU中,只有12个是丰富类群,中等和稀有分别为161和2870个。DNBSEQ和 NovaSeq揭示了几乎相同的丰富和中等微生物类群,表明这两个类群的测序平台效应相对较低(图 4A)。然而,对于稀有微生物类群,在NovaSeq平台观察到了更多的平台特异类群。在2870个稀有OTU中,仅在一个平台检测到有913个(32%),而其中889个分类多样的稀有OTU仅在NovaSeq平台检出,远高于DNBSEQ的24个(图 4A)。
NovaSeq平台特异检测到的物种多样的稀有类群会导致样本Alpha和Beta多样性结果受到影响。不同测序平台Alpha多样性比较发现NovaSeq平台的 Chao I 指数显著降低,但其系统发育多样性更高,表明其特有的稀有类群比DNBSEQ覆盖了更广泛的物种类群。NovaSeq特异的稀有OTU频率分析表明,超过30%的OTU仅在一个样本中出现(图 5B),这与观察到的更高的Beta多样性和更高的整合群落多样性一致(图 4A)。此外,NovaSeq 特异检测到的6个门(Armatimonadetes, BRC1, Chloroflexi, Genmatinonadetes, Deinococcus thermus, candidate division WPS-2)均不是牛瘤胃系统中常见的微生物类群(图 4B)。为了进一步评估NovaSeq特异的稀有类群是真实的稀有类群还是在测序过程中引入的假阳性,我们计算了每个稀有类群与瘤胃液理化参数(包括NH4+、乙酸盐、丙酸盐、丁酸盐和异丁酸盐等)之间的相关性。假设是牛瘤胃中真实的稀有类群,其应该与其宿主的发酵条件相关,与理化参数相关的概率高于随机引入的假阳性。与NovaSeq平台检测到的稀有类群相比,DNBSEQ平台检出的稀有类群相关性比例始终较高,且与每个理化参数显著相关,进一步表明了NovaSeq平台检测到的假阳性稀有类群比例可能高于DNBSEQ。
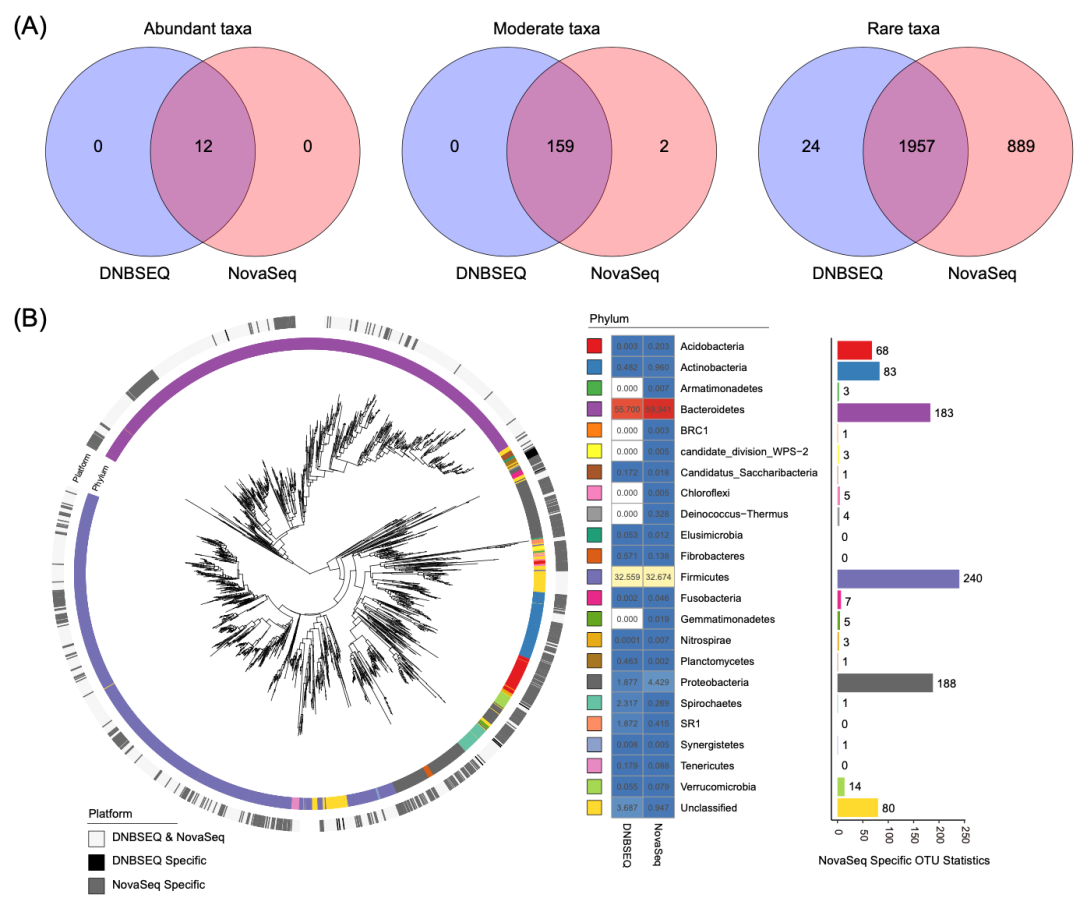
图4 不同测序平台牛瘤胃微生物群落结构特征。
■ 标签跳跃会导致微生物群落构建机制研究产生偏差
不同生态系统中微生物群落的构建同时受到随机和确定性过程的影响,每个过程控制不同生态系统中微生物群落组成的不同部分。了解微生物的群落构建机制对于研究微生物组如何干预宿主健康至关重要。然而,大量假阳性/假阴性稀有类群的产生如何影响我们对群落构建机制的解释与认识仍不清楚。我们使用Sloan中性选择模型以及零模型(null assembly model)对两个测序平台上得到牛瘤胃微生物群落组成结果进行分析,以确认随机或确定性过程是否在牛瘤胃微生物的群落构建过程占主导地位,以及不同的测序平台是否会导致相似或不同的构建机制。我们发现微生物群落的Sloan中性模型中性拟合系数较低(DNBSEQ R2 = 0.321;NovaSeq R2 = 0.360),且两个测序平台的结果较一致(图 5A)。此外,估算迁移率 m 广泛用于评估群落中个体的随机损失将被从整合群落中的漂移物种所取代的概率,这与群落中的繁殖率相反。我们发现 DNBSEQ-G400 的 m 值较 NovaSeq 6000 大(DNBSEQ m = 0.221;NovaSeq m = 0.079),表明了瘤胃微生物群落在同一个环境中不同奶牛之间存在潜在的交流。
同时我们对每个平台测序结果计算了β-NTI指数 (β-nearest taxon index),以进一步区分牛瘤胃微生物群落构建中的确定性和随机过程。通过分别统计随机性选择 (|β-NTI| < 2)、变量选择 (β-NTI ≥ 2) 和同质化选择 (β-NTI ≤ -2) 解释的群落构建过程(图 5C),我们发现与Sloan的中性模型拟合结果一致,两个测序平台的β-NTI分布表明牛瘤胃微生物群落构建同时受到随机和确定性过程的影响。与DNBSEQ相比,NovaSeq平台的β-NTI值分布范围更广,并且仅在NovaSeq结果中观察到同质化选择的迹象(图 5C),而将NovaSeq数据特异检测的OTU删除后得到了与DNBSEQ平台相似的β-NTI值分布模式(图 5C)。
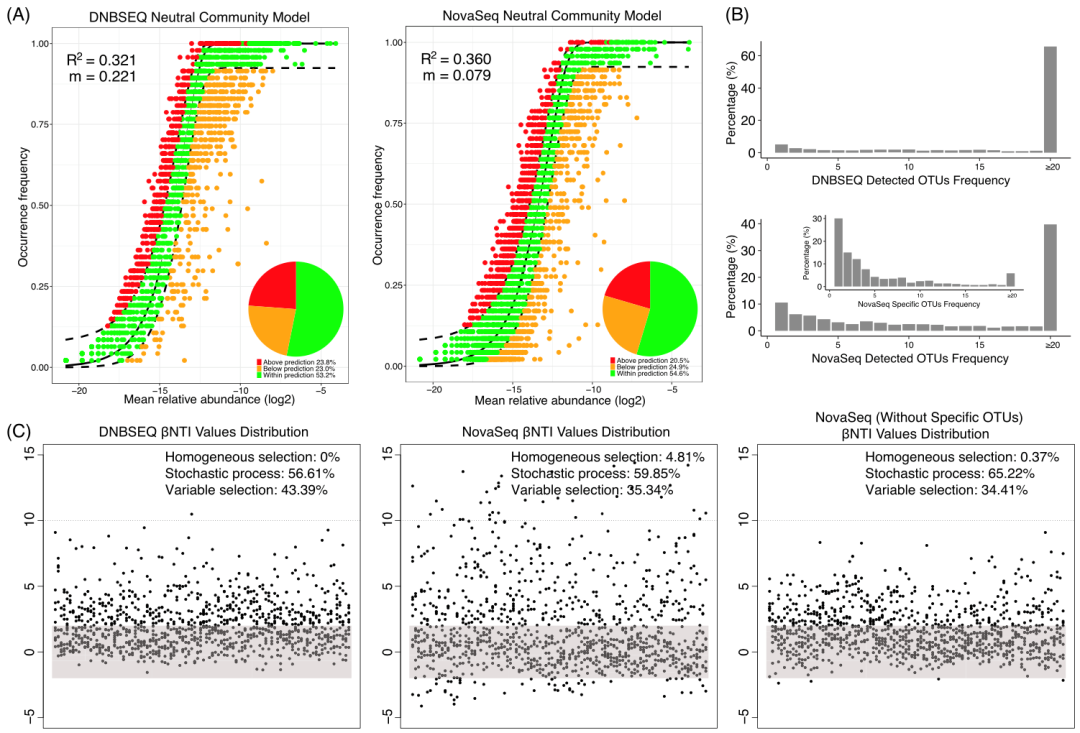
图5 牛瘤胃微生物群落构建机制解析。
■ 不同测序平台导致微生物互作网络核心物种产生差异
已有很多研究通过样本间微生物相对丰度的相关系数来推断微生物之间的相互作用。网络图的结构或拓扑特征可以为复杂的微生物间相互作用和共现模式提供宝贵的见解,并可用于识别在群落中核心物种等发挥重要作用的微生物。因此,我们对假阳性的产生是否会导致对微生物相互作用的误导甚至错误解释进行了评估。对每个测序平台瘤胃微生物群落构建共现相关性互作网络,发现稀有类群对每个网络的节点均贡献超过了90%,展示了稀有类群在牛瘤胃中潜在的重要生态作用(图 6A)。两个平台的互作网络节点度均服从幂律分布,显示出无标度网络的性质,然而与基于DNBSEQ平台的网络相比,基于NovaSeq平台的微生物网络集成度较低,随机移除节点后其网络稳定性显著降低(图 6B)。虽然来自毛螺菌科(Lachnospiraceae)、梭菌目(Clostridiales)、拟杆菌目(Bacteroidales)和普雷沃氏菌(Prevotella)等微生物在两个平台中都认为是核心物种(图 6C),但DNBSEQ鉴定的两个核心物种(木糖假丁酸弧菌Pseudobutyrivibrio xylanivorans,瘤胃琥珀酸菌Succiniclasticum ruminis)均有报道其在瘤胃系统中发挥重要的生态作用,而这两个物种并未在NovaSeq的互作网络中的占据核心位置。相反,NovaSeq平台鉴定出的几个核心物种在DNBSEQ平台上属于低关联度或未被检测到(如Nocardia coeliaca,Otu0244)。其中 Nocardia coeliaca 是一种好氧革兰氏阳细菌,并不是牛瘤胃中常见的微生物,我们使用专门设计的引物对 Nocardia coeliaca 进行了PCR验证,仅能获得弱阳性克隆,其Sanger测序结果与 Nocardia coeliaca Otu0244 相似性较低,证实了瘤胃样本中并不存在Nocardia coeliaca。
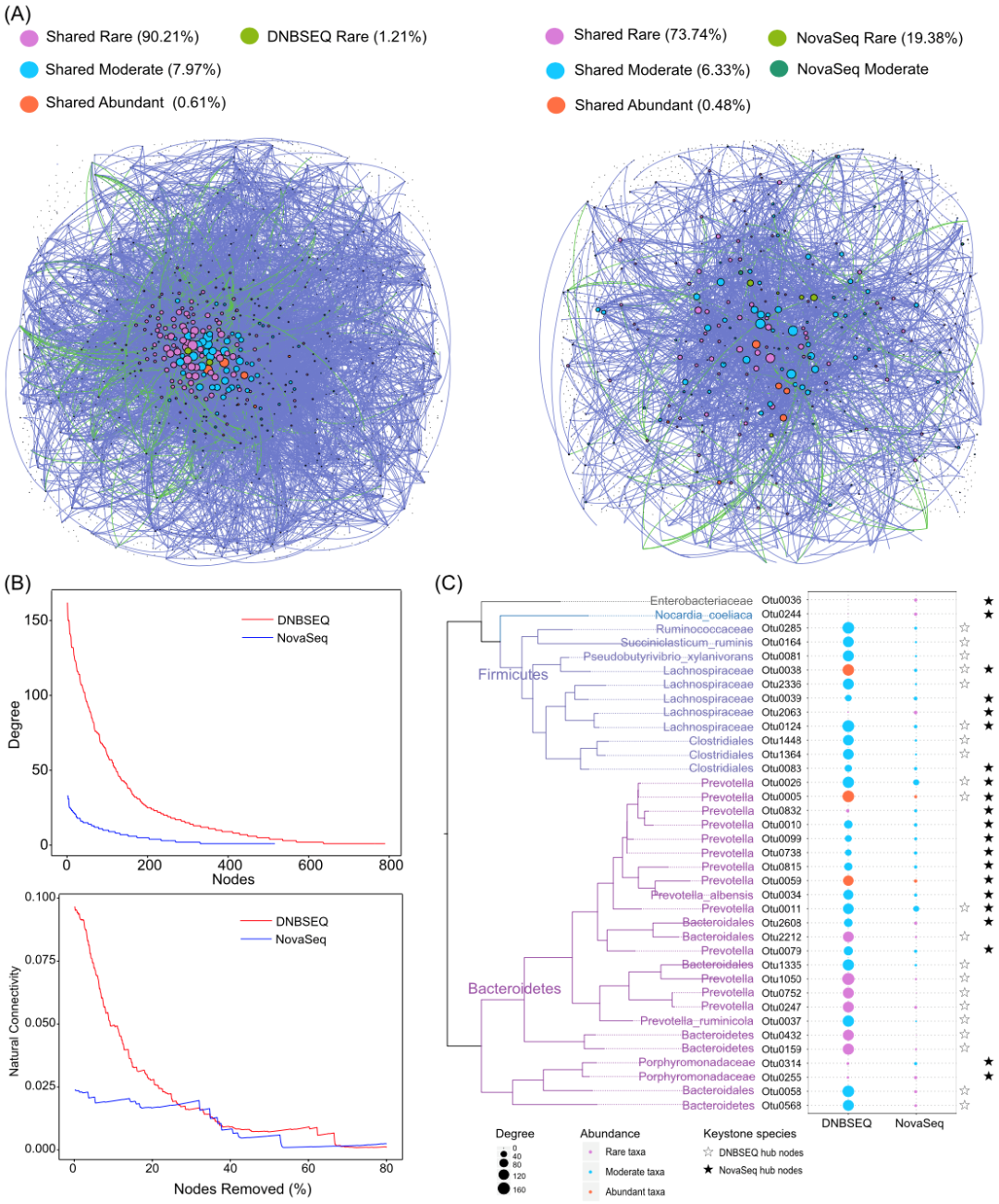
图6 不同测序平台牛瘤胃微生物群落互作网络比较。
05 研究结论
在扩增子研究中,虽然样本间标签跳跃不会对相对高丰度的类群产生显著影响,但它可能对稀有群落的分析产生偏差,包括群落组成、多样性、物种相互作用网络、群落构建机制等。从实验的任何过程中都有引入潜在的污染,包括提取、PCR扩增、文库构建、测序及其它操作。由于标签跳跃以随机方式发生,我们假设适当的技术重复和重复间详细的交叉验证可以减少部分假阳性的OTU,但当我们使用NovaSeq测序平台时,即使对同一批次样本进行技术重复测序也可能存在真实稀有类群的丢失及假阳性稀有微生物的引入,不能保证所有真实的稀有类群都被一致检测到。因此当重点关注稀有微生物类群时,应谨慎的分析其假阳性及假阴性的可能。同时正确的设置阳性和阴性对照,包括空白提取试剂盒,以及数据处理过程中适当的质量控制和生物信息学算法,也可用于消除潜在的污染。此外,建议使用具有低标签跳跃率和足够测序深度的适当测序平台来提高稀有类群检测和下游生物生态机制解释的准确性。尤其在关注稀有微生物类群的研究中,高标签跳跃(index hopping)率不仅会通过样本交叉污染引入假阳性稀有类群,同时真实的稀有类群也可能通过标签跳跃丢失造成假阴性结果,因此我们建议使用不同的测序技术对扩增子测序结果进行交叉验证。
附DNBSEQ平台已发表扩增子文章:
1. Jia, Y. et al. Sequencing introduced false positive rare taxa lead to biased microbial community diversity, assembly, and interaction interpretation in amplicon studies. Environmental Microbiome 17, 43 (2022). https://doi.org/10.1186/s40793-022-00436-y
2. Wei, Y. et al. The microbial diversity in industrial effluents makes high-throughput sequencing-based source tracking of the effluents possible. Environmental Research 212, 113640 (2022).https://doi.org/10.1016/j.envres.2022.113640
3. Anslan, S. et al. Highly comparable metabarcoding results from MGI-Tech and Illumina sequencing platforms. PeerJ 9, e12254 (2021). https://doi.org/10.7717/peerj.12254
4. Yang, C. et al. Efficient COI barcoding using high throughput single-end 400 bp sequencing. BMC Genomics 21, 862 (2020). https://doi.org/10.1186/s12864-020-07255-w
5. Sun, X. et al. Efficient and stable metabarcoding sequencing data using a DNBSEQ-G400 sequencer validated by comprehensive community analyses. Gigabyte 2021, 1-15 (2021). https://doi.org/10.46471/gigabyte.16
6. Chen, J. et al. Revealing an Invasion Risk of Fish Species in Qingdao Underwater World by Environmental DNA Metabarcoding. Journal of Ocean University of China 20, 124-136 (2021). https://doi.org/10.1007/s11802-021-4448-2
7. Zou, K. et al. eDNA metabarcoding as a promising conservation tool for monitoring fish diversity in a coastal wetland of the Pearl River Estuary compared to bottom trawling. Sci Total Environ 702, 134704 (2020). https://doi.org/10.1016/j.scitotenv.2019.134704
8. Yang, X., Song, X., Hallerman, E. & Huang, Z. Microbial community structure and nitrogen removal responses of an aerobic denitrification biofilm system exposed to tetracycline. Aquaculture 529 (2020). https://doi.org:10.1016/j.aquaculture.2020.735665
9. Li, C. et al. A survey of the sperm whale (Physeter catodon) commensal microbiome. PeerJ 7, e7257 (2019). https://doi.org/10.7717/peerj.7257
排版|乔维钧